
생화학분자생물학회입니다.
다중 오믹스 분석(Multi-Omics Analysis) – 질병 연구의 새로운 접근
작성자
관리자작성일자
2017-12-01조회수
4613다중 오믹스 분석
(Multi-Omics Analysis)
– 질병 연구의 새로운 접근

이연수
국립암센터 임상의학연구부
yslee2@ncc.re.kr
1. 서론
“다중오믹스” 분석은 유전체(Genome), 전사체(Transcriptome), 단백체(Proteome), 대사체(Metabolome), 후성유전체(Epigenome), 지질체(Lipodome) 등 다양한 분자 수준에서 생성된 여러 데이터들의 총체적이고 통합적인 분석을 의미한다. 이러한 총합적 분석은, 초고속(High-Throughput) 분자생물학적 분석 기술들의 발전과 가격 측면에서의 급격한 부담 감소 및 컴퓨터 산업의 발전에 따른 정보 처리 능력의 비약적 발달에 따라 가능하게 되었다. 단일 분석에 비해서 다중오믹스 분석이 거의 혁명적이라고 여겨지는 것은, 다양한 데이터들의 총합적 분석이 줄 수 있는 정보가 단일 분석에 비해 훨씬 많아서, 암 등 복잡한 원인으로 발생하는 질병의 연구에 중요한 정보를 제공하며, 진단이나 치료법 개발에 있어서 보다 큰 기여를 할 수 있다고 여겨지기 때문이다. 단일의 ‘~체(-omics)’ 분석 기술은 단지 하나의(예를 들면 DNA, RNA, Protein 등) 분자 수준만을 보여주기 때문에, 얻을 수 있는 정보가 매우 제한적인 반면에 다중오믹스 분석은 다수의 ‘~체’ 정보를 통합하여 보다 글로벌한 정보를 제공한다.[1][s1] 따라서, 다중오믹스 분석은, 서로 독립적인 층(Layer)에서의 오믹스 분석 정보들을 입체적으로 통합하여(Multi-Layer) 생물학적 메커니즘을 보다 명확히 보여줄 수 있기 때문에, 어떤 현상(예: 질병)의 결과로 나타나는 변화(Consequential Changes)가 아니라 원인이 되는 변화(Causative Changes)를 더 잘 밝혀낼 수 있다고 믿어지고 있다.
다중오믹스 분석은 크게 유전형, 표현형 그리고 환경적인 요인에 기반을 둔 접근으로 구분할 수 있다 (1). 유전형 기반 다중오믹스 분석은 질병의 위험과 연관되어 있는 좌위(Locus)를 찾는 것을 목적으로, 먼저 전장유전체연관성분석(Genome Wide Association Study, GWAS)을 수행하고, 관련된 유전자 후보군을 도출한 다음, 유전체(변형 데이터)나 전사체(발현 수준 데이터)를 사용하여 후보군을 검증한다. 표현형 기반 다중오믹스 분석은 질병과 임상적 요인들 그리고 오믹스 기반 데이터들 사이의 연관성을 분석하여 정보를 얻는데, 질병의 진행에 대한 생물학적 이해가 더 많이 이루어질 수 있다는 장점이 있다. 환경요인 기반의 다중오믹스 분석은 미생물체(Microbiome), 유전체 또는 대사체 데이터와 같은 오믹스 데이터들을 통합하여 얻은 데이터를 가지고 흡연이나 다이어트 같은 환경적인 요인과의 연관성을 추정한다 (그림 1).
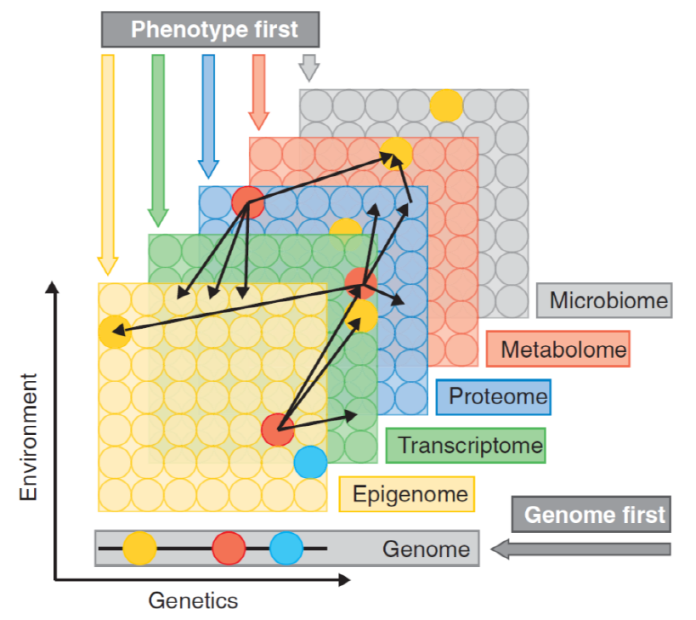
그림 1. 다중오믹스데이터 종류와 질병 연구를 위한 접근 방법 (1)
2. 본론
2-1. 다중오믹스 분석에서 고려해야 할 점
인간의 유전체는 매우 복잡하고, 다층 수준에서 조절되고 있어 하나가 아닌 다양한 오믹스 방법에 의해서만이 전체적인 정보를 얻을 수 있다. 예를 들면, 폐암의 진단과 치료에 있어서 필요한 정보들만 고려해 본다고 해도, 단순한 Mutation(Insertion과 Deletion을 포함하는), Copy Number Variation, Fusion Gene, RNA와 Protein 수준의 발현 변화 등 다층 수준의 정보가 필요하다. 이러한 정보들은 각각이 별개의 것이 아니며 복잡한 시스템 안에서 상호 작용을 하여 질병(또는 형질)을 나타낸다. 따라서 이러한 다층 수준의 오믹스 정보들을 총합하여 생물학적 시그너처를 찾고 이를 통해 질병에 유의한 정보를 도출하는 것은 매우 도전적인 일이다.
대량의 비교와 맞춤형 통계분석 및 시간과 인력과 경제적 부담이 큰 다중오믹스 연구에 있어서, 연구의 디자인 단계에서 고려해야 할 점들은 다음과 같다:
1) 질병 인과관계의 복잡성(Complexity of Disease Etiology) – 단순히 하나의 유전자가 연관된 것이 아닌, 복합질병(Complex Disease)의 경우 다수의 조절유전자(Modifier Genes)들과 다수의 요인(환경 시간 등)이 영향을 미치기 때문에 다수의 오믹스 데이터가 여러 요인에 걸쳐서(예를 들면 다수의 시간대) 수집될 필요가 있다.
2) 데이터 생산 및 이후의 분석 과정 – 오믹스 데이터는 시료의 특성에 따르는 다수의 플랫폼을 통해 얻어지기 때문에 이를 통합할 수 있는 방법에 대한 고려가 필수적이다. 컴퓨터 파이프라인을 이용할 수밖에 없는 오믹스 데이터의 통합은 매우 큰 용량의 저장공간을 필요로 하며, 서로 다른 분석 장비를 사용하여 생산된 오믹스 데이터를 전처리하여 공통으로 사용할 수 있게 만드는 것은 쉽지 않다. 이러한 문제들은 데이터 통합 자체를 위해서도 중요하지만, 통합된 데이터를 이용하여 도출된 정보(예를 들면 환자와 정상인 간에 다른 정보)가 통합과정에서 나온 기술적인 오류로 인해 오염될 수 있는 가능성을 차단하기 위해 반드시 풀려야 하는 것들이다. 아직까지 데이터 통합에 대한 황금률(Gold Standard)는 없지만, 다수의 방법들이 개발되고 있으며[2] 데이터 수집 및 기술적 문제에 대한 해법들이 많이 제기되고 있다.
3) 얻고자 하는 파워(예측력 등)와 그에 연관된 샘플의 수 – 오믹스 분석은 대개 통계적인 접근을 동반하는데, 그때 연관성을 발견할 수 있는 파워는 샘플의 크기에 크게 좌우된다. 질병 연구에서 필수적으로 따라다니는 교란 변수들(식습관, 환경 등)을 고려하지 못한 샘플 수는 위양성(False Positive) 결과로 귀결될 수밖에 없으며 이는 다중오믹스 분석과 같이 대규모 데이터를 다루어야 하는 연구에서는, 연구를 시작하기 전에 매우 중요하게 고려되어야 하는 사항이다.
4) 모델 동물에 대한 다중오믹스 분석 – 질병의 연구에 있어서, 인간의 시료에 대한 접근은 매우 제한적이며 특히 다중오믹스 분석에 필요한 양과 질이 충분히 확보된 시료는 흔하지 않다. 이런 제한점을 극복하기 위해, 동물모델에 대한 분석이 제시되고 있으며, 이들 동물모델의 장점은 특히 재현성(Reproducibility), 환경 등 다수 요인들에 대한 제어, 반복 실험의 가능성, 표현형에 대한 정확한 제어, 가설에 대한 실험적 검증의 가능성 등에 있어서 매우 크다고 여겨진다. 정확히 제어되고 반복된 실험에서 얻어진 다중오믹스 데이터는, 제한적으로 얻어진 인체 시료의 데이터와 비교 분석됨으로써 보다 폭넓고 정확한 연구결과를 제공할 수 있다.
2-2. 다중오믹스 분석 방법
다중오믹스 분석을 위한 통합 방법은 최근에 주목받고 있는 정밀 의료 분야에도 큰 이슈가 되고 있다. 임상 시료의 다양성과 질병의 복잡성을 고려할 때, 다중오믹스는 필수불가결한 분석 방법이 되고 있으며 임상에서 필요한 정확하고 재현성 있는 결과를 내기 위한 통합 방법의 개발은 급속도로 발전하고 있다. 현재까지 개발되고 활용되고 있는 분석 방법의 개요를 아래에 정리해보았다 (세부 내용은 표1) (2).
비지도 데이터 통합법(Unsupervised Data Integration): 인풋 데이터에 레이블을 하지 않고 클러스터링을 하는 방법으로 데이터 자체의 특성을 이용해서 소그룹(Cluster)을 얻고 이를 생물학적 특성과 연계시킨다. 1) Matrix Factorization Methods(Joint Non-negative Matrix Factorization(NMF), iCluster, iCluster+, Joint and Individual Variation Explained(JIVE), Joint Bayes Factor), 2) Correlation-Based Analysis, 3) Bayesian Methods(Multiple Dataset Integration(MDI)-Patient-Specific Data Fusion(PSDF), Bayesian Consensus Clustering(BCC), COpy Number and EXpression In Cancer(CONEXIC)), 4) Network-Based Methods(PAthway Representation and Analysis by DIrect Reference on Graphical Models(PARADIGM), Similarity Network Fusion(SNF), Lemon-Tree), 5) Multiple Kernel Learning 및 Multi-Step Analysis (Regularized Multiple Kernel Learning Locality Preserving Projections(rMKL-LPP), CNAmet, In-Trans Process Associated and Cis-Correlated(iPAC)) 등 다섯 가지로 분류할 수 있다.
지도 데이터 통합법(Supervised Data Integration): 비지도 데이터 통합법과는 반대로, 샘플에 이미 아는 대로 레이블을 하고 데이터를 훈련시키는 방법으로, 레이블된 샘플의 정보를 이용하여 데이터의 패턴을 찾고 이 패턴을 이용하여 레이블되지 않은 데이터를 분석한다. 1) Network-Based methods(jActiveModules), 2) Multiple Kernel Learning(Semidefinite Programming/Support Vector Machine(SDP/SVM)), 3) Multi-Step Analysis(Multiple Concerted Disruption (MCD), Anduril) 등 세 가지로 분류할 수 있다.
준-지도 데이터 통합법(Semi-Supervised Data Integration): 위의 두 가지 방법의 중간적인 방법으로, 객체 기반의 유사성 네트워크를 만들고 레이블된 객체의 관계를 통해서 레이블되지 않은 객체를 분석하며, 대부분은 그래픽 기반으로 되어 있다. 대표적으로 GeneticInterPred과 같은 방법이 있다.
아직까지 대다수의 다중오믹스 데이터 통합, 분석 방법은 장점과 단점을 함께 가지고 있어, 하나만 써서 원하는 결과를 쉽게 도출할 수 있는 수준이 되지는 못하고 있다. 이들 분석 방법의 개발은 컴퓨터 성능의 증가와 밀접한 연관이 있으며 생물학적 지식의 축적 및 정확하고 풍부한 임상 데이터의 결합과도 결부되어 있다. 특히, 다중 인종이나 다중 환경에서 생산된 매우 이질적인 데이터의 통합 분석은 아직까지 극히 부족한 상태로 보다 적극적인 개발이 필요하다.
표 1. 데이터 통합 기술들 (2)
.png)
.png)
2-3. 암 연구에서 다중오믹스의 최근 결과
최근 셀(Cell)지에 발표된 연구는 (3) 단백체, 인지질 단백체 및 유전체 데이터를 통합하여 분석하는 다중오믹스 연구를 통해 난소암의 생물학적 이해에 매우 큰 진전을 이루어냈다. 이 연구를 보도한 기사에 따르면 (4), 임상단백체종양분석컨소시엄(The Clinical Proteomic Tumor Analysis Consortium, CPTAC[3])의 연구진은, 고등급 장액성 난소암(High-Grade Serous Carcinomas, HGSCs) 환자들의 종양조직에서 단백체를 분석하여, 이전에 얻었던 유전체, 전사체 데이터와 통합하는 시도를 했다. 연구자들은 종양 샘플에서 9,600여 개의 단백질들을 찾아냈으며, 모든 종양 샘플에서 나타난 3,586개의 단백질들을 선별해냈다. 이 결과에 더해, 연구진은 복제수변이(Copy Number Alterations, CNAs)가 단백질의 양에 미치는 효과를 찾아내서, 서로 다른 염색체에 위치한 네 개 부위의 CNV가 200여 개 이상의 단백질의 양을 변화시키는 데 영향을 미쳤다는 것을 밝혔다. 이때, 대부분의 전사체들의 양은 변하지 않아서, CNA가 단백질에 미치는 영향이 번역 후(Post-Translational) 수준에서 이루어졌음을 암시했다. CNA에 의해 영향을 받는 단백질들은, 특히 암의 진행과 연관된 세포 이동이나 침윤 및 면역 조절에 관련된 것들이 많았으며, 그중 몇몇 단백질들은 암과의 연관성이 이미 밝혀진 것들이었다. 임상데이터의 분석은 이들, 네 개의 CNA 부위에 의해 조절되는 단백체 특징(Proteome Signature)이 환자의 생존과 강한 연관이 있음을 나타냈으며, 이는 CNA의 트랜스(Trans) 활성에 의해 조절되는 단백질들 중에, 암의 나쁜 진행과 연관된 여러 기전에 관여하고 있는 것들이 다수 있음으로 또다시 확인되었다.
연구자들은 또한, PARP(Poly(ADP-ribose) Polymerase) 저해제 치료와 연관되고 생존율을 높이는데 관련된, 상동재조합 결핍(Homologous Recombination Deficiency, HRD)과 연관된 단백질들을 살펴보았다. 여기서, HRD를 가진 환자와 가지지 않은 환자를 구별하는, 대략 30여 개의 단백질들로 구성된 네트워크가 밝혀졌고, 더 나아가 HRD 환자들에게서 이중나선 손상 복구(Double-Stranded Break Repair) 기전에 관여하는 히스톤탈아세틸효소(Histone Deacetylase 1, HDAC1)가 높아져 있으면서 H4(Histone 4)의 아세틸화가 줄어든, 번역 후 변형(Post-Translational Modification) 수준에서의 차이도 나타났다. 그리고 69명의 암조직으로부터 인산화 펩티드에 대해 분석하고 데이터베이스(Pathway Interaction Database, PID)를 이용해 경로를 추적한 후, 단기간 생존자와 장기간 생존자의 인산화 단백질, 단백질 또는 전사체 수준에서의 데이터에 근거하여 활성을 평가해 보았다. 흥미롭게도 인산화 단백질 데이터를 썼을 때는 단기간 생존자에게서 15개의 경로가 유의하게 활성이 높아진 것으로 나오는 반면, 전사체 데이터를 썼을 때는 단 하나의 경로만이 유의하게 활성화된 것으로 나타났다. 이러한 결과는 유전체와 전사체, 단백체들이 서로 어떻게 다르게 층화되어 있는지를 보여주는 좋은 예로, 다중오믹스가 임상적 결과를 보다 정확하게 예측하고 분석할 수 있다는 것을 명확하게 보여준다.
2-4. 단일 세포(Single-Cell) 수준의 다중오믹스 분석
다중오믹스 분석은, 또한 정밀의학 (5) 및 단일 세포에 대한 연구 방법으로 최근 주목을 받고 있다. 단일 세포에서 유전체, 후성유전체, 전사체 또는 단백체를 분석해내는 방법은 이미 활발히 이용되고 있으며[4] 서로 다른 세포 간의 비교나 질병의 분석 등에서 좋은 결과들을 도출하고 있다. 하지만 단일 오믹스 데이터는 세포의 상태를 일부만 보여줄 수 있으며 이는 보다 총합적인 분석이 가능한 다중오믹스 분석으로 해결이 가능할 것으로 여겨지고 있다. 단일 세포 다중오믹스 분석은 특히, 세포 내의 다양한 변이들과 단백질 및 표현형 발현 간의 다이나믹한 연관관계를 명확하게 보여줄 수 있어 세포의 발달 및 질병의 경과의 연구에 필수적인 정보를 제공할 것이다 (6).
단일 세포 다중오믹스 분석에서 기술적으로 가장 앞서있는 것은, 유전체와 전사체의 병행 분석이다. 이는 유전체 변이와 전사체 발현 및 표현형 간의 상관관계를 밝혀줌으로써 유전체 변이의 기능적인 역할을 보여줄 수 있다. 최근 개발된 기술은 gDNA-mRNA Sequencing(DR-seq) 방법으로 단일 세포 용해물(Lysate) 내의 gDNA와 mRNA를 분리하지 않고 동시에 라이브러리를 만들어 염기서열을 분석하는 방법이다 (7). 또한 gDNA와 mRNA를 먼저 분리하여 분석하는 G&T-seq(Genome and Transcriptome Sequencing) 방법도 있다 (8). 두 방법은 전자는 gDNA와 mRNA의 손실이 최소화될 수 있는 장점이 있는 반면, 후자는 두 분자들 사이의 오염이 최소화될 수 있다는 장점이 있다.
최근에는 여기에 더해 후성유전체 변이와 전사체 분석을 연결시키려는 다중오믹스 연구가 시도되어 Single-Cell Bisulfite Sequencing(scBS-seq)과 Single-Cell Reduced Representation Bisulfite Sequencing(scRRBS-seq)을 Single-Cell RNA-seq과 결합시켰는데, 초기 시도로서는 scMT-seq(Single-Cell Methylome and Transcriptome Sequencing) 방법을 쓴 결과가 있다 (9). 또한 유전체, 후성유전체 및 전사체 데이터까지 연계시키는 시도도 있었으며 (10), 질량분석방법의 발달과 더불어, 단일 세포 수준의 전사체와 단백체 동시 분석도 이루어질 수 있다는 것이 보고되었다 (11).
이러한, 단일 세포 수준의 다중오믹스분석은 세포계통을 밝히는 데 특히 유용하며, 암과 같은 질병에서 발병 전후 및 치료 전후와 재발 전후 등 세포의 다양성과 계통을 밝혀 진단과 치료에 직접적으로 활용할 수 있는 분야에 핵심적인 기술로 자리매김할 것으로 보인다 (그림 2).
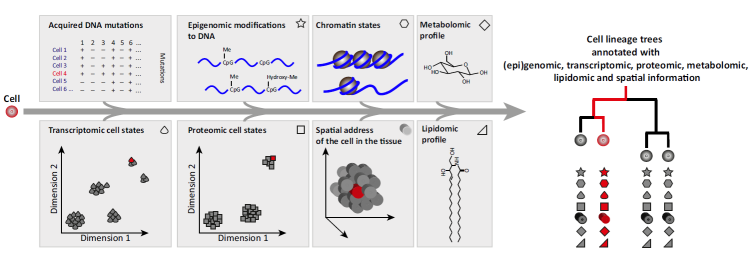
그림 2. 단일 세포를 이용한 다중오믹스분석. 암과 같은 질병에서 매우 중요한 Cell Lineage 연구에 유용한 정보를 제공한다 (6).
3. 결어
유전체, 전사체, 단백체, 후성유전체 등 다수의 오믹스 데이터를 총합하는 다중오믹스 분석은, 아직까지는 이론이나 개념으로 구성된 부분이 많이 남아 있음에도 불구하고 매우 빠른 발전 속도를 보이고 있다. 특히 단일 세포 분석과 관련된 기술들이 급속도로 발전하고 있어서(Pacific Biosciences, Oxford Nanopore 등) 다중오믹스 분석이 임상을 포함하여 다양한 분야에서 단기간 내에 많은 응용이 가능할 것으로 낙관되고 있다. 가까운 미래에 다중오믹스 분석 연구는 정적이고 개별적인 것이 아닌, 생물학적 역동성이 충분히 반영된 세포와 조직, 개체에 대한 정보들을 제공할 것이며 이는 생명체의 근본적인 이해를 위한 위대한 도약이 될 것이다.
참고문헌
1. Hasin, Y., Seldin, M., Lusis, A. (2017) Multi-omics approaches to disease. Genome Biol 18, 83-98.
2. Huang, S., et al. (2017) More is Better: Recent Progress in Multi-Omics Data Integration Methods, Front Genet 8, 84.
3. Zhang, H., et al. (2016) Integrated Proteogenomic Characterization of Human High-Grade Serous, Ovarian Cancer. Cell 166, 755-765.
4. Waldron, D. (2016) A multi-layer omics approach to cancer. Nat Rev Genet 17, 436–437
5. Rotroff, D.M., Motsinger-Reif, A.A. (2016) Embracing Integrative Multiomics Approaches. Int J Genomics 2016, 1715985
6. Macaulay, I.C., et al. (2017) Single-Cell Multiomics: Multiple Measurements from Single Cells, Trends Genet 33, 155-167
7. Dey, S.S., et al. (2015) Integrated genome and transcriptome sequencing of the same cell. Nat Biotechnol 33, 285-289
8. Macaulay, I.C., et al. (2016) Separation and parallel sequencing of the genome and transcriptome of single cells using G&T-seq. Nat Protoc 11, 2081-2103
9. Angermueller, C., et al. (2016) Parallel single-cell sequencing links transcriptional and epigenetic heterogeneity. Nat Methods 13, 229-232
10.Hou, Y., et al. (2016) Single-cell triple omics sequencing reveals genetic, epigenetic, and transcriptomic heterogeneity in hepatocellular carcinomas. Cell Res 26, 304-319
11.Frei, A.P., et al. (2016) Highly multiplexed simultaneous detection of RNAs and proteins in single cells, Nat Methods 13, 269-275
[1] 예를 들면, 장님들의 코끼리 만지기에 비유해 볼 수 있다. 여섯 명의 장님들이 각각 코끼리의 다리 네 개(Genome, Transcriptome, Proteome, Metabolome)와 머리(Epigenome)와 꼬리(Lipidome)를 만지고 의논한다고 해도 코끼리 자체를 설명할 수는 없다. 이들이 만진 각각은 코끼리의 부분에 불과하기 때문에 서로 통합되어 유기적으로 설명될 때만 코끼리를 나타내게 된다. 또한 어떤 데이터를 어떤 것과 비교 분석하는 가는 결과에 매우 중요한 영향을 주는데, 코끼리의 예를 들면, 앞 다리와 뒷 다리의 데이터는 서로 매우 유사하고 비교할 수 있지만 (피부, 근육, 형태 등), 꼬리와는 (피부 등 다수의 연관성은 있지만) 직접 비교하기 어렵다. 오믹스데이터의 경우도, 층화(Layer)되어 있는 데이터들이 서로 멀어질수록 단순 비교가 어렵고 연관성이 떨어지게 되며 비교 방법의 구축도 어렵다.
[2] 구체적인 방법은 본 고 뒤쪽의 ‘다중오믹스 분석 방법’ 및 표 1 참조
[3] 미국 NIH 산하의 국립암연구소(The National Cancer Institute, NCI)의 주도로 이루어 지고 있는 전국적인 연구로, 대규모 유전단백체연구를 통해 종양의 분자적기반을 이해하고자 하는 노력으로, 2011년 대장암, 유방암과 난소암을 파일럿으로 하여 시작했으며, 현재는 The Applied Proteogenomics OrganizationaL Learning and Outcomes (APOLLO) Network와 The International Cancer Proteogenome Consortium (ICPC)로 확장되었다. 데이터 포탈에는 2017년 11월 현재 7.48 TB의 단백체 데이터가 있으며, 전세계 2만여명의 사용자가 현재까지 261.2TB의 데이터를 내려받기하여 사용하고 있다. (데이터는 공공에서 무상으로 사용할 수 있다.)
[4] 관련하여, 생화학회 Webzine/소식지 8월호에 ‘종양연구에서 단일 세포 분석(Single Cell Analysis in Cancer Biology)’ (주경민, 성대의대) 분석 기사가 실려있다.
첨부파일