
생화학분자생물학회입니다.
전사 단계에서의 자식작용 조절 (Transcriptional Regulation of Mammalian Autophagy)
작성자
관리자작성일자
2017-04-01조회수
835전사 단계에서의 자식작용 조절
(Transcriptional Regulation of Mammalian Autophagy)


신지현 조동형
경희대학교 경희대학교
동서의학대학원 동서의학대학원
노인학과 노인학과
jhshin8759@gmail.com dhcho@khu.ac.kr
서론
2016년 노벨생리의학상은 자식작용(자가포식, Autophagy) 연구의 선구자로, 자식작용 조절 유전자와 기본 원리를 밝힌 일본 도쿄공업대(Tokyo Institute of Technology)의 오스미 요시노리(Ohsumi Yoshinori) 명예교수에게 돌아갔다. Autophagy는 그리스어로 ‘자기’를 뜻하는 auto와 ‘포식’을 뜻하는 phagein을 합친 말로 ‘스스로 먹는다’는 뜻으로, 자식작용은 필요가 없어지거나 손상된 세포소기관 또는 비정상적인 단백질 등을 오토파고좀(Autophagosome)을 통한 분리와 최종적으로 라이소좀(Lysosome) 의존적으로 분해하는 ‘청소부’ 역할을 한다. 이와 같이 자식작용은 우리 몸에서 항상 일어나는 생리적 현상이며, 노화되거나 손상된 세포 구성물질을 제거함으로써 항상성 유지에 중요한 기능을 담당하며, 평상시에는 매우 낮은 수준을 유지하고 있다.
특히, 자식작용은 영양결핍(Starvation), 저산소증(Hypoxia), 감염(Infection), ER 스트레스(ER stress) 등과 같은 다양한 스트레스 조건에서는 높은 활성을 나타내며, 과도한 스트레스를 줄여 세포의 항상성 유지와 세포생존에 중요한 기능을 담당한다 (1). 따라서 자식작용 조절의 이상은 퇴행성뇌질환, 암, 당뇨, 감염성 질환 등 많은 종류의 질병과 연관되어 있고, 몇몇 자식작용 활성 조절 약물은 이러한 질병의 동물 모델에서 성공적인 결과를 보여주고 있다 (2). 특히, 알츠하이머(Alzheimer’s), 헌팅턴(Huntington’s) 질병이나 파킨슨(Parkinson’s) 등과 같은 퇴행성 뇌질환은 신경세포에서 독성 단백질 응집체의 축적과 관련되어 있는데, 자식작용의 활성은 이러한 세포 내 독성물질을 제거하는 역할을 하기 때문에 관련 질환의 새로운 치료제 개발 가능성을 제시하고 있다 (3, 4).
초기의 자식작용 연구는 유전자 발현 제어에 따른 자식작용 조절보다는 역동적인 세포막 재배열과 ATG(Autophagy-Related Gene) 유전자 동정과 단백질의 번역 후 변형(Post-Translational Modification)에 중점을 두었다. 실제로 자식작용이 핵을 제거한 세포(Enucleated Cell)에서도 유도될 수 있다는 발견은, 핵에서 일어나는 반응이 자식작용 과정에서 그렇게 중요하지 않다는 가정으로 이어졌다 (5). 하지만 1999년 효모를 이용한 초기 연구에서 질소 결핍에 의한 자식작용 유도는 몇 분 안에 ATG 유전자의 전사 과정을 증가시킨다는 결과를 보여주었다 (6). 특히, Transcription Factor EB(TFEB)가 많은 자식작용 연관 유전자를 조절한다고 보고된 2011년 이후부터, 자식작용의 전사 조절에 대한 보고가 급속히 증가하고 있다. 비록 현재까지 20여개 이상의 전사조절인자(Transcription Factor)가 자식작용 활성에 관여되어 있다고 보고 되었으나, 전사조절인자의 작용 기전에 대한 연구는 상당히 미흡하고 자식작용의 중요한 조절자로써의 역할에 대해서는 여전히 많은 연구가 진행되어야 한다. 따라서 본론에서는 포유류(Mammalian) 자식작용과 자식작용 타겟 유전자에 관련된 세포 내 전사조절인자에 중점을 두어 최신 연구결과를 알아보고자 한다.
본론
2-1. TFEB 와 ZKSCAN3 – The Master Autophagy Regulator
라이소좀 Biogenesis의 마스터(Master) 조절자로 알려진 Transcription Factor EB(TFEB)은 라이소좀 생성 뿐만 아니라 자식작용 관련 여러 유전자 발현을 조절하는 주요 인자로 보고되었다 (7). TFEB의 과발현 만으로도 자식작용 연관 단백질들을 빠르게 유도할 수 있다. 영양 충분 조건에서 TFEB는 mTOR에 의해 인산화되면서 세포질에 존재하지만, 영양결핍과 Rapamycin 처리와 같은 자극에 의해 자식작용이 활성화된 후에는 mTOR가 억제되고 TFEB가 탈인산화 되어 핵으로 빠르게 이동한다 (그림 1) (8). 핵으로 이동한 TFEB는 많은 자식작용 연관 유전자의 프로모터(Promotor)에 직접적으로 결합하여 ATG4, ATG9B, Microtubule-Associated Protein 1 Light Chain 3B(MAP1LC3B), UV Radiation Resistance Associated Protein(UVRAG), WD-Repeat Domain Phosphoinositide Interacting Protein(WIPI)을 포함하는 자식작용과 Lysosome의 흐름(Autophagic Flux)을 조절하는 중요한 단백질의 발현을 유도한다 (그림 1). TFEB는 Lysosomal Biogenesis의 주요 조절인자로, 자식작용 연관 유전자 발현 조절뿐만 아니라 Autophagic Flux 등 여러 자식작용 과정의 다양한 단계를 조절하는 능력을 보여주고 있다 (7). TFEB의 과발현은 설치류 모델에서 단백질 응집체에 관련된 질병의 치료 효과를 보여준다. 예를 들면, TFEB 과발현은 α-Synuclein의 독성을 없애고, α-Synuclein의 과발현에 의해 유도된 파킨슨 질병의 쥐 모델에서 도파민 신경세포를 보호한다 (3). 또한 TFEB 과발현은 Misfolded Polyglutamine-Expanded(poly Q) Huntingtin 단백질의 제거와 X-Linked 척추와 전구 근 위축(Bulbar Muscular Astrophy)을 일으키는 Androgen 수용체 돌연변이를 제거함으로써 독성을 개선한다 (4, 9). 이와 같이, 결론적으로, TFEB의 전사 활성에 의한 자식작용 조절은 다양한 병리학적 모델에 중요한 역할을 하는 것으로 보인다.
최근 TFEB의 전사 조절 카운터파트(Transcriptional Counterpart)로 Zinc-Finger Protein with KRAB and SCAN Domains 3(ZKSCAN3)은 Unc-51-Like Autophagy Activating kKnase1(ULK1)과 MAP1LC3B를 포함하는 많은 자식작용 연관 유전자의 전사를 억제한다 (10). TFEB와 달리, 자식작용이 유도되면 ZKSCAN3는 핵에서 세포질로 이동하여 TFEB에 의한 타켓 유전자의 전사 활성이 일어날 수 있도록 해준다. ZKSCAN3 발현 억제만으로도 자식작용이 유도하고 ZKSCAN3 과발현은 자식작용을 억제할 수 있다 (그림 1) (10). 이처럼 TFEB와 ZKSCAN3는 서로 다른 Shuttling 과정을 통해 자식자용 연관 유전자 발현을 조절한다 (그림 1).
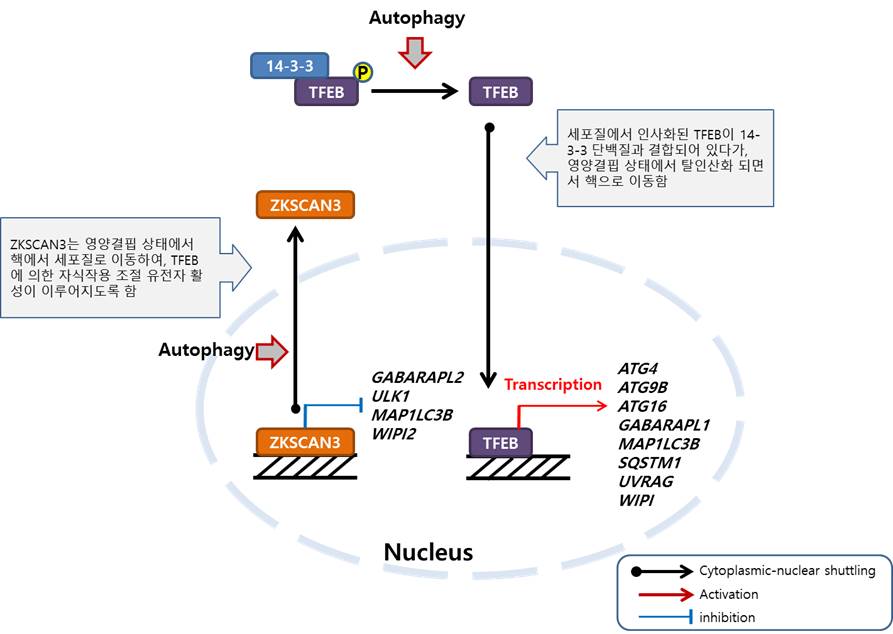
그림 1. 전자 조절인자 TFEB 와 ZKSCAN3에 의한 자식작용 관련 유전자 발현조절 양상
2-2. 저산소증과 자식작용 – Well Studied But Still Enigmatic
여러 병리 조건과 연관되어 있는 저산소증(Hypoxia)은 대표적인 자식작용 유도 조건으로 사용되어 왔으며, 많은 문헌들에서 저산소증에 의한 ATG 유전자의 전사 조절을 보고하였다. 저산소증 Master 조절인자인 Hypoxia-Inducible Factor 1 Alpha(HIF1α)에 의한 BNIP3와 BNIP3L의 발현 조절은 여러 논문에서 조명되었다 (11). BNIP3는 beclin1/ATG6에 의한 B-cell Lymphoma 2(BCL2)의 억제를 없앰으로 자식작용의 양성 조절자(Positive Regulator) 역할을 한다. 흥미롭게도, 저산소증의 정도에 따라 서로 다른 전사 조절인자가 자식작용을 조절하는 것으로 보인다 (그림 2). 예를 들어, 중등도 저산소증에서는 HIF1α가 BNIP3 전사를 활성화시키는 반면, 심한 저산소증은 Activating Transcription Factor 4(ATF4)가 관여하는 것으로 보고되었다 (11). ATF4는 저산소증 상태에서 MAP1LC3의 프로모터에서 Cyclic AMP Response Element(CRE) 결합 부위에 직접 결합함으로써 MAP1LC3B의 전사를 유도하며, ULK1은 ATF4에 의해 활성화 되는 반면, ATG5는 ATF4에 의해 조절되는 DNA Damage Inducible Transcript 3(DDIT3)의 전사 유도를 통해 간접적으로 활성된다 (12).
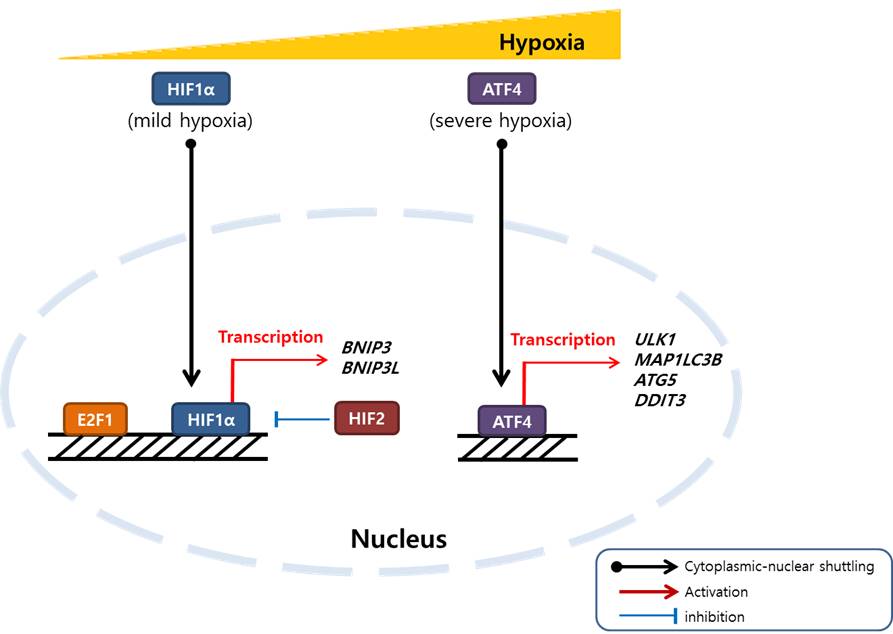
그림 2. 저산소증에서 전사조절인자 HIF1α와 ATF4에 의한 자식작용 관련 유전자 발현조절 양상
2-3. The FOXO Family – Location Matters
다양한 발생학적 과정과 조직 항상성을 비롯한 여러 생리적 기능과 관련 있는 FOXO(Forkead Box Protein) 단백질들은 자식작용과 관련되어 최초로 보고된 전사조절인자 중 하나이다. TFEB와 같이 FOXOs는 인산화에 의해 조절되고 ATG4, ATG12, BECN1, BNIP3, MAP1LC3B, ULK1, VPS34 (PIK3C3), GABARAPL1을 포함하는 많은 자식작용 연관 유전자의 발현을 유도하기 위해 핵으로 이동한다 (13). 근육과 심장에서 FOXK1의 핵과 세포질 사이의 이동은 mTOR과 Chromosomal Maintenance 1(CRM1)에 의존하고, 아미노산 결핍에 의한 mTOR 억제는 염색질(Chromatin)로부터의 분리를 야기한다 (14). 추가적으로 FOXO1의 핵으로의 이동은 ATG5, ATG14, PIK3C3의 전사 활성과 관련 있다 (15). Transcription Factor X-Box-Binding Protein 1(XBP1)은 FOXO1 활성과 분해의 중요한 또 다른 조절인자이다. Spliced XBP1은 Beclin1의 프로모터 부분에 직접 결합하여 Splice Isoform에 따라 자식작용 활성인자이거나 억제인자로 역할을 할 수 있다 (16). TFEB와 달리 FOXO1은 세포질 내에서 자식작용 연관 단백질과 직접 결합하여 자식작용 유도자(Inducer)로써 기능을 하기도 한다 (17). 따라서, FOXO Family 의 구성원은 세포 내 위치에 따라 자식작용 유도자와 억제자(Repressor)로 역할을 할 수도 있으며, 이러한 특징은 p53도 같이 갖는다.
2-4. p53 – Between Cell Survival and Death
p53은 대표적인 암 억제 유전자로 세포주기와 세포사멸을 조절하며, 유전자 돌연변이를 예방하여 게놈(Genome)의 안정성 보존에 중요한 역할을 한다. 많은 선행 연구결과들은 p53이 TORC1을 억제하여 자식작용을 활성시킨다고 알려졌다. 하지만 몇몇의 연구에서 cytoplasmic p53은 자식작용의 억제자로써 기능을 보여주고 있으나, 자식작용 억제에 대한 조절기전은 많이 알려져 있지 않다 (18). 핵 안에서의 p53의 기능은 유전체 연구에서 조사되었으며, ATG2, ATG4, ATG7, ATG10, ULK1 등 여러 자식작용 연관 유전자들의 프로모터 부위에 p53이 결합된다 (19). DNA 손상이나 종양 유전자의 활성 등에 의한 자식작용 유도는 p53을 활성화 시키고 자식작용을 증가시킨다. 더욱이 p53 Family 단백질인 p63과 p73도 자식작용 연관 유전자를 타겟 유전자로 가지고 있어, 특정 조건에서 p53의 손실에 의한 자식작용 조절을 보완할 수 있다 (19). 한편, N-Terminally Truncated p63 Isoform(ᐃNp63α) 의 인산화된 형태인 p-ᐃNp63α는 ULK1, ATG5, ATG7을 포함하는 다양한 자식작용 유전자의 프로모터에 결합할 수 있을 뿐만 아니라 miRNA의 전사를 통해 간접적으로 자식작용을 조절하는 반면, p73은 mTOR에 의해 억제되고 자식작용 유도제인 Rapamycin에 의해 유도된다 (20). 따라서, p53 Family 구성원은 다양한 자극에 따라 여러 가지 자식작용 연관 유전자를 조절하는 가능을 가지고 있다. 주목할 만하게도 세포의 생사를 결정하는 p53의 주요 보조 단백질인 E2F1도 자식작용 연관 유전자의 중요한 전사 조절인자로 기능을 하고 있다.
2-5. E2F1 과 NF-kB – Competing For The Spotlight
세포주기 활성 조절 주요 단백질로 알려진 E2F1 전사조절인자의 활성은 자식작용을 유도시키고, 반대로 E2F1 단백질의 감소는 자식작용을 억제한다 (21). E2F1은 ULK1, MAP1LC3A, BINP3 등의 자식작용 연관 유전자를 타겟으로 가지며 또한 ATG5의 전사를 간접적으로 조절하는 것으로 알려졌다 (21). 또한, Nuclear Factor Kappa-Light-Chain-Enhancer of Activated B Cells(NF-κB)는 E2F1 프로모터의 결합을 억제함으로써 BNIP3의 Transactivation을 위한 분자적 스위치로써 알려져 있다 (22). 따라서, E2F1은 BNIP3의 전사를 활성시킴으로써 자식작용을 유도하는 반면에 NF-kB는 이 transactivation을 억제하는 상호 경쟁하는 관계이다. 이 두 자식작용 조절인자 사이의 또 다른 관련성은 E2F1에 의한 NF-κB의 억제자인 IκB의 안정화에 기인한다 (21). 그럼에도 불구하고, NF-κB는 또한 Beclin1과 Sequestosome-1(SQSTM1)을 포함하는 자식작용 연관 유전자를 유도시킨다고 알려져 있다. 최근에는 IAPs(Apoptosis Inhibitory Proteins)와 XIAP(X-Linked Inhibitor of Apoptosis Protein)는 NF-κB의 활성을 통해 Beclin1 전사를 증가시키므로 자식작용을 유도한다고 보고되었다 (23). 주목할만한 내용은, ATG 유전자 또는 자식작용의 유도를 위해 단백질의 전사 활성이 항상 필요한지 여부는 명백하지 않다. 예를 들어 전사 활성 영역(Transcriptional Activity Domain)이 결여된 E2F1은 여전히 자식작용을 유도할 수 있다 (24).
2-6. Jun – Activated By Various Stresses
c-Jun N-Terminal Kinase(JNK) 경로는 사이토카인 및 산화 스트레스 등 환경적인 스트레스에 의해 활성화 되어 다양한 세포신호 전달에 관여한다. 자식작용 또한 세포 스트레스에 대해서도 활성화되기 때문에 두 경로 사이의 연결은 예상치 못한 일이 아니다. 최근 영양결핍에 대한 반응으로 자식작용의 Vesicular Trafficking에 관여하는 Annexin A2(ANXA2)가 JNK-Jun 경로에 의해 전사 조절되는 것으로 보고되었다 (25). ANXA2 과발현 자체가 자식작용을 유도하기 때문에, JNK-Jun-ANXA2 Transcriptional Program은 영양결핍에 반응하여 자식작용을 조절하는 중요한 과정으로 보인다 (25). Jun은 ANXA2 뿐만 아니라, Beclin 1과 MAP1LC3B 발현 조절에도 관여하는 것으로 알려져 있다 (12).
2-7. FXR-PPARα-CREB Axis
최근 핵 수용체와 자식작용 사이의 주목할만한 관련성이 한국과학자가 주도한 두 개의 논문에 의해 알려졌다 (26, 27). 이들 논문은 자식작용 조절 유전자들이 자식작용을 유도하는 절식 조건(Fasting Condition)과 비교하여 섭식 조건(Feeding Condition)하에 간에서 Farnesoid X Receptor(FXR)에 의해 직접적으로 억제된다는 같은 결과를 제시하였지만 다른 조절 기전을 제안하였다. Fasting Transcriptional Activator인 CRE-Cinding Protein(CREB)은 ATG7, ULK1 및 TFEB를 비롯한 자식작용 유전자를 활성화 시킨다. 이들은 다른 방식으로 FXR에 의해 억제되며, CREB와 CREB-Regulated Transcription Coactivator 2(CRTC2) 사이의 기능적 복합체(Functional Complex)를 파괴한다 (26). 다른 한편으로, FXR과 다른 영양소 감지 조절제인 Peroxisome Proliferation Factor-Activated Receptor α (PPARα)의 반대되는 역할을 설명했다. PPARα는 금식으로 활성화되며 특정 DNA 결합 부위(DR1)를 FXR과 공유하며, FXR이 활성화되면 PPARα의 결합이 억제된다 (27). PPARα와 CREB 복합체가 MAP1LC3A와 ATG7 유전자를 다르게 조절한다는 결과에 의해 두 가지 메커니즘이 함께 작용할 수 있다.
2-8. 그 외의 전자 인자
앞서 언급된 전사인자 이외에도 많은 전사 인자들이 자식작용의 모든 단계에서 자식작용 연관 유전자의 전사 활성과 관련되어 있다. 이러한 전사조절인자의 대부분은 자식작용 유도에 의해 세포질에서 핵으로 이동하며 기능적 전위(Functional Translocation)라고 부른다. 놀라운 예로서, Proteasome 26S Subunit Non-ATPase 10(PSMD10)은 최근 아미노산 결핍 상태에서 핵으로 이동하는 것으로 보고되었으며 이것의 전사를 유도하기 위해 ATG7 프로모터에 전사 인자 Heat Shock Factor Protein 1(HSF1)과 결합한다 (30). 주목할 만하게, 간에서 자식작용과 Lysosome 흐름 및 자식작용 관련된 유전자의 발현은 24시간 주기 리듬(Circadian Rhythm)을 따르는 것처럼 보인다. 따라서 아미노산 결핍에 의해 자극 될 수 있는 CCAAT/Enhancer Binding Protein β(C/EBPβ)는 MAP1LC3A, MAP1LC3B를 비롯한 여러 ATG 유전자를 활성시킨다 (31). 최근 연구는 MAP1LC3A의 프로모터에서 CREs의 존재를 강조하였고, 실제로 GABARAPL1 프로모터에 CREB1 결합은 GABARAPL1 발현에 필요하다. 자식작용의 다양한 유도자에 의해 활성하고 자식작용 관련 유전자의 프로모터와 결합하는 전사 인자에 대한 연구는 증가하고 있고 자식작용 관련 유전자의 전사 조절을 통해 자식작용을 조절하는 것으로 나타나는 포유류 전사 인자의 목록은 표. 1에서 볼 수 있다.
결론 (Perspective)
전사 단계의 자식작용 조절 연구는 TFEB에 대한 연구로 인해 폭발적으로 증가했다. TFEB, Jun 및 FOXO3와 같은 전사 인자에 대한 연구는 단일 전사 인자의 변경된 활성이 자식작용을 유도하거나 억제하는데 충분할 수 있음을 보여주었다. 비록 관련 연구가 크게 증가하고 있으나 자식작용을 조절하는 전사인자의 종류는 아직 많이 밝혀져 있지 않은 편으로 향후, 다른 여러 전사인자에 의한 주요 자식작용 유전자 발현 조절이 다양한 스트레스 반응과 자식작용과 연관된 병리적 작용을 조절할 가능성이 남아 있다. 또한 이러한 유전자 발현 조절은 특정 스트레스에 의한 세포 특이적 또는 세포 선택적 자식작용 활성을 나타낼 수도 있을 것이다. 유전자 발현 조절과 관련하여 주목할 만한 부분은 지난 몇 년 동안 자식작용에 대한 핵의 역할은 전사 인자의 조절에 국한되지 않고, Epigenetic 제어, microRNA 및 핵과 세포질 사이의 주요 자식작용 단백질의 이동을 포함한다는 것이 밝혀졌다. 더욱이 최근 밝혀지고 있는 자식작용 조절 주요 유전자들의 Epigenetic 조절은 유전자 발현이 매우 복잡하게 제어되고 있음을 보여준다. 따라서 앞으로, 이러한 요인들이 어떻게 서로 다른 자식작용 결과를 가능하게 하기 위해 상호 연결되는지는 이 분야에서 가장 흥미로운 질문 중 하나이다. 또한 앞으로 자식작용의 전사 조절인자에 대한 세포 유형 특이성(Cell-Type Specificity) 측면으로 접근하는 것이 중요할 것이다.

표. 1 포유류에서 주요 자식작용 유전자를 조절하는 전사인자
참고문헌
1. Klionsky, D.J., Abdelmohsen, K., Abe, A., Abedin, M.J., Abeliovich, H., Acevedo, Arozena, A. et al. (2016)
Guidelines for the use and interpretation of assays for monitoring autophagy (3rd edition). Autophagy 12: 1-222
2. Jiang, P., Mizushima, N. (2014) Autophagy and human diseases. Cell Res. 24: 69-79.
3. Decressac, M., Mattsson, B., Weikop, P., Lundblad, M., Jakobsson, J. and Bjö rklund, A. (2013) TFEB-mediated
autophagy rescues midbrain dopamine neurons from α-synuclein toxicity. Proc Natl Acad Sci. USA 110: E1817-E1826.
4. Tsunemi, T., Ashe, T. D., Morrison, B. E., Soriano, K. R., Au, J., Roque, R. A. V.,
Lazarowski, E. R., Damian, V. A., Masliah, E. and La Spada, A. R. (2012) PGC- 1α rescues Huntington’s disease
proteotoxicity by preventing oxidative stress and promoting TFEB function. Sci Transl Med. 4: 142ra97.
5. Tasdemir, E., Maiuri, M. C., Galluzzi, L., Vitale, I., Djavaheri-Mergny, M., D’Amelio, M., Criollo, A.,
Morselli, E., Zhu, C., Harper, F. et al. (2008) Regulation of autophagy by cytoplasmic p53.
Nat Cell Biol. 10: 676-687.
6. Kirisako, T., Baba, M., Ishihara, N., Miyazawa, K., Ohsumi, M., Yoshimori, T., Noda, T. and Ohsumi, Y. (1999)
Formation process of autophagosome is traced with Apg8/Aut7p in yeast. J Cell Biol. 147: 435-446.
7. Settembre, C., Di Malta, C., Polito, V. A., Garcia Arencibia, M., Vetrini, F., Erdin, S., Erdin, S. U., Huynh, T.,
Medina, D., Colella, P. et al. (2011) TFEB links autophagy to lysosomal biogenesis. Science 332: 1429-1433.
8. Martina, J. A., Chen, Y., Gucek, M. and Puertollano, R. (2012) MTORC1 functions as a transcriptional regulator of
autophagy by preventing nuclear transport of TFEB. Autophagy 8: 903-914.
9. Cortes, C. J., Miranda, H. C., Frankowski, H., Batlevi, Y., Young, J. E., Le, A., Ivanov, N., Sopher, B. L.,
Carromeu, C., Muotri, A. R. et al. (2014) Polyglutamine-expanded androgen receptor interferes with TFEB to elicit
autophagy defects in SBMA. Nat Neurosci. 17: 1180-1189.
10. Chauhan, S., Goodwin, J. G., Chauhan, S., Manyam, G., Wang, J., Kamat, A. M. and Boyd, D. D. (2013) ZKSCAN3
is a master transcriptional repressor of autophagy. Mol Cell. 50: 16-28.
11. Pike, L. R. G., Singleton, D. C., Buffa, F., Abramczyk, O., Phadwal, K., Li, J.-L., Simon, A. K., Murray, J. T.
and Harris, A. L. (2013) Transcriptional up-regulation of ULK1 by ATF4 contributes to cancer cell survival.
Biochem J 449: 389-400.
12. Füllgrabe J, Ghislat G, Cho DH, Rubinsztein DC. (2016) Transcriptional regulation of mammalian autophagy at a
glance. J Cell Sci. 129: 3059-3066.
13. Mammucari, C., Milan, G., Romanello, V., Masiero, E., Rudolf, R., Del Piccolo, P., Burden, S. J., Di Lisi, R., S
andri, C., Zhao, J. et al. (2007) FoxO3 controls autophagy in skeletal muscle in vivo. Cell Metab. 6: 458-471.
14. Bowman, C. J., Ayer, D. E. and Dynlacht, B. D. (2014) Foxk proteins repress the initiation of starvation-induced
atrophy and autophagy programs. Nat Cell Biol. 16: 1202-1214.
15. Xiong, X., Tao, R., DePinho, R. A. and Dong, X. C. (2012) The autophagy-related gene 14 (Atg14) is regulated by
forkhead box O transcription factors and circadian rhythms and plays a critical role in hepatic autophagy and
lipid metabolism. J Biol Chem. 287: 39107-39114.
16. Margariti, A., Li, H., Chen, T., Martin, D., Vizcay-Barrena, G., Alam, S., Karamariti, E., Xiao, Q.,
Zampetaki, A., Zhang, Z. et al. (2013) XBP1 mRNA splicing triggers an autophagic response in endothelial cells
through BECLIN-1 transcriptional activation. J Biol Chem. 288: 859-872.
17. Zhao, Y., Yang, J., Liao,W., Liu, X., Zhang, H.,Wang, S.,Wang, D., Feng, J., Yu, L. and Zhu, W. G. (2010)
Cytosolic FoxO1 is essential for the induction of autophagy and tumour suppressor activity.
Nat Cell Biol. 12: 665-675.
18. Scherz-Shouval, R.,Weidberg, H., Gonen, C.,Wilder, S., Elazar, Z. and Oren, M. (2010) p53-dependent regulation of
autophagy protein LC3 supports cancer cell survival under prolonged starvation.
Proc Natl Acad Sci. USA 107: 18511-18516.
19. Kenzelmann Broz, D., Spano Mello, S., Bieging, K. T., Jiang, D., Dusek, R. L., Brady, C. A., Sidow, A. and
Attardi, L. D. (2013) Global genomic profiling reveals an extensive p53-regulated autophagy program contributing
to key p53 responses. Genes Dev. 27: 1016-1031.
20. Huang, Y., Guerrero-Preston, R. and Ratovitski, E. A. (2012) Phospho- DeltaNp63alpha-dependent regulation of
autophagic signaling through transcription and micro-RNA modulation. Cell Cycle. 11: 1247-1259.
21. Polager, S., Ofir, M. and Ginsberg, D. (2008) E2F1 regulates autophagy and the transcription of autophagy genes.
Oncogene. 27: 4860-4864.
22. Shaw, J., Yurkova, N., Zhang, T., Gang, H., Aguilar, F.,Weidman, D., Scramstad, C., Weisman, H. and
Kirshenbaum, L. A. (2008) Antagonism of E2F-1 regulated Bnip3 transcription by NF-kappaB is essential for basal
cell survival. Proc Natl Acad Sci. USA 105: 20734-20739.
23. Lin, F., Ghislat, G., Luo, S., Renna, M., Siddiqi, F. and Rubinsztein, D. C. (2015) XIAP and cIAP1 amplifications
induce Beclin 1-dependent autophagy through NFkappaB activation. Hum Mol Genet. 24: 2899-2913.
24. Garcia-Garcia, A., Rodriguez-Rocha, H., Tseng, M. T., Montes de Oca-Luna, R., Zhou, H. S., McMasters, K. M.
and Gomez-Gutierrez, J. G. (2012) E2F-1 lacking the transcriptional activity domain induces autophagy.
Cancer Biol Ther. 13: 1091-1101.
25. Moreau, K., Ghislat, G., Hochfeld, W., Renna, M., Zavodszky, E., Runwal, G., Puri, C., Lee, S., Siddiqi, F.,
Menzies, F. M. et al. (2015) Transcriptional regulation of Annexin A2 promotes starvation-induced autophagy.
Nat Commun. 6: 8045.
26. Seok, S., Fu, T., Choi, S. E., Li, Y., Zhu, R., Kumar, S., Sun, X., Yoon, G., Kang, Y., Zhong, W. et al. (2014)
Transcriptional regulation of autophagy by an FXRCREB axis. Nature 516: 108-111.
27. Lee, J. M., Wagner, M., Xiao, R., Kim, K. H., Feng, D., Lazar, M. A. and Moore, D. D. (2014) Nutrient-sensing
nuclear receptors coordinate autophagy. Nature 516: 112-115.
28. Yan, S., Yang, X., Chen, T., Xi, Z. and Jiang, X. (2014) The PPARγ agonist Troglitazone induces autophagy,
apoptosis and necroptosis in bladder cancer cells. Cancer Gene Ther. 21: 188-193.
29. Qin, H., Tan, W., Zhang, Z., Bao, L., Shen, H., Wang, F., Xu, F. and Wang, Z. (2015). 15d-prostaglandin J2
protects cortical neurons against oxygen–glucose deprivation/reoxygenation injury: involvement of inhibiting
autophagy through upregulation of Bcl-2. Cell Mol Neurobiol. 35: 303-312.
30. Luo, T., Fu, J., Xu, A., Su, B., Ren, Y., Li, N., Zhu, J., Zhao, X., Dai, R., Cao, J. et al. (2015)
PSMD10/Gankyrin induces autophagy to promote tumor progression through cytoplasmic interaction with ATG7 and
nuclear transactivation of ATG7 expression. Autophagy. [Epub ahead of print] doi:10.1080/15548627.2015. 1034405.
31. Ma, D., Panda, S. and Lin, J. D. (2011) Temporal orchestration of circadian autophagy rhythm by C/EBPbeta.
EMBO J. 30: 4642-4651.
첨부파일