
생화학분자생물학회입니다.
세포 이미징 및 단백체 분석이 가능한 BRD4 Inhibitor 기반 Minimalist 광가교 저분자 화합물 개발
작성자
관리자작성일자
2018-04-01조회수
795세포 이미징 및 단백체 분석이 가능한 BRD4 Inhibitor 기반 Minimalist 광가교 저분자 화합물 개발
A Suite of “Minimalist” Photo-Crosell Imaging and Chemical Proteomics: Case Study with BRD4 Inhibitors
Angewandte Chemie International Edition 56:11816-11821, 2017
|
|
|
|
이준석
|
장세영
|
연구배경
생체 내 약물의 Target 단백질을 규명하기 위한 방법은 크게 Cell Phenotypic Screening, in silico 기반 분석, Affinity Chromatography 기반 질량분석이 있다. 이중 Affinity Chromatography 기반 분석방법은 in vitro 상에서 직접적인 Drug Target Clue를 제공하는 실험 기법이지만 Inhibitor와 단백체 간의 Non-Specific Binding으로 인한 False Positive 및 Missing Target에 대한 문제가 항상 동반되며 더불어 Inhibitor를 Immobilized 시킬 수 있는 다양한 Matrix의 부재로 인해 그 사용이 제한적인 문제를 가지고 있다. 이러한 문제점을 극복하기 위해 살아있는 세포의 특정 단백질만 선택적으로 레이블링 할 수 있는 Biocompatible Chemical Tag를 통한 단백체 레이블링이 최근 주목받고 있다. 이 방법은 특정 단백체에 레이블링 할 수 있도록 디자인된 화합물로 인한 증가된 선택성과 함께 살아있는 세포에 적용이 가능하다는 점으로 인해서 Off-Target, Protein Pathway 분석이 가능하여 기존보다 더 발전된 Target Validation이 가능하다는 장점을 가진다 (1).
이를 위한 Biocompatible Chemical Tag 합성 전략은 크게 3가지로 나뉘는데 1) 약물의 특성을 저해하지 않도록 Linker의 크기 및 구조를 디자인해 약물에 컨쥬게이션 해야 하고 2) 대부분 Non-Covalent 결합인 Drug-Protein 간 결합을 자외선 조건에서 Covalent 결합으로 변환할 수 있도록 하는 Photocrosslinker 도입 3) Covalent 결합된 단백질을 선택적으로 레이블링 하여 다양한 Cellular Study에 응용될 수 있도록 하는 Click Chemistry 기반 Affinity Handle 도입이다 (그림 1-A). 이렇게 합성된 Chemical Tag를 세포 내에 처리 후 얻어지는 단백체 질량분석 결과는 약물의 Off-Target 및 생체 내 약물-단백질 상호작용과 이로 인해 발생되는 단백질 시그널 Pathways 분석에 매우 중요한 정보를 제공한다.
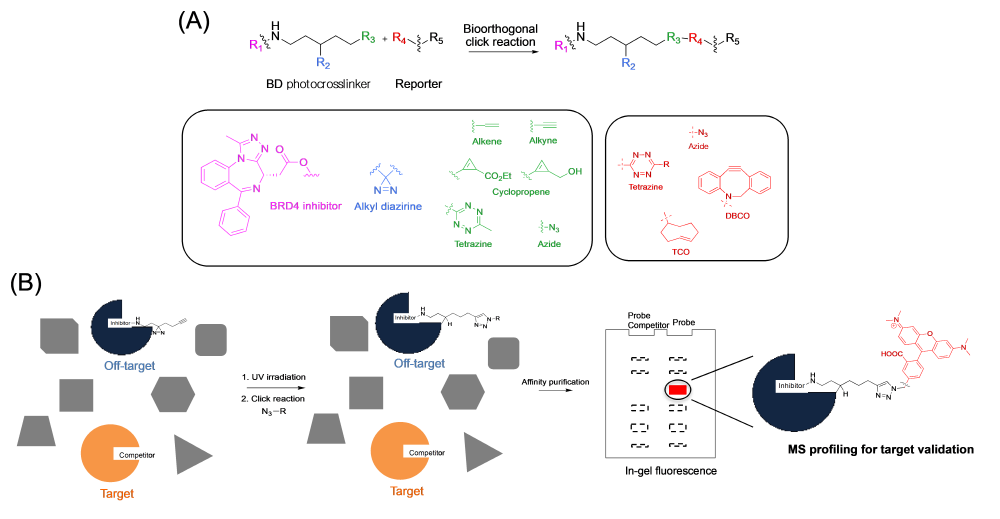
그림 1. (A) 연구에서 사용된 Biocompatible BD Chemical Tag 의 구조로 BD 화합물은 BRD4 Inhibitor, Photocrosslinker, Clickable Handle 3가지 Part로 구성되어 있다. 각 작용기의 역할은 BRD4 Inhibitor의 BRD4 Inhibition, Diazirine Photocrosslinker의 BRD4 단백체와의 공유결합 유도, Click Handle과 Reaction Pair인 형광 Reporter와의 Ligation이며 이러한 특징으로 인해 BD 화합물은 이미징 및 질량분석 단백체 분석에 모두 사용이 가능하다. (B) BD Chemical Tag를 이용한 BRD4 Off-Target 도출 방법에 대한 요약 설명으로 Wild Type BRD4 Inhibitor 와 BD의 경쟁 반응을 통해 BRD4가 아닌 Off-Target 분자에 공유결합된 화합물은 Click 반응과 함께 형광 Scanning 할 수 있게 된다. 이렇게 특정하게 레이블링 된 단백질은 후에 질량분석을 통해 어떠한 단백질인지 프로파일링 하게 된다.
AfBP를 구성하는 중 대표적인 Photocrosslinker인 Alkyl Diazirine은 350nm 영역에서 매우 반응성이 큰 Carbene 중간체 형태로 변형되어 주변부 아미노산과의 Proximal Reaction을 통해 앞서 설명한 Non-Specific 공유결합을 할 수 있다. 특히 다른 Linker와 다르게 사이즈가 작아 반응간섭이 적고 반응 중간체인 Carbene 반감기가 Nanosecond로 매우 빨라 적은 S/N Ratio로 단백체에 레이블링 할 수 있는 장점이 있다 (그림 1-A) (2). 최근 이러한 Photocrosslinker를 사용해 특정 단백질에 대해서 정교하고 반응성이 높은 화학반응이 유도될 수 있도록 하는 High-Quality Chemical Tag 개발이 활발히 이뤄지고 있으며 특히 동일한 모체에 서로 다른 구조의 Photocrosslinker가 Whole Proteome 레벨에서 Target Validation에 어떠한 영향을 주는지에 관련된 연구 결과도 보고가 되고 있다 (3).
Bromodomain and Extra-Terminal(BET) 계열 단백질은 Histone의 Acetylated Lysine (Kac) Reader이다. 특히 BET은 최근 암치료에 있어서 중요한 Target 단백질로 주목을 받고 있다. 그중 BRD4 는 혈액종양이나 고형 종양에서 많이 발견되고 있고, 특정 Gene Loci에 대한 BRD4의 결합은 c-Myc, BCL2와 같은 Transcription Protein Complex를 유도하여 암세포의 성장 및 사멸에 관여하는 것으로 밝혀졌다 (4). 이에 착안하여 2010년 옥스퍼드 대학 James Brander 연구팀은 단백질 구조 분석을 통해 Triazole-Diazepine Ring 구조체 기반 BRD4 Inhibitor인 JQ1을 발표해 Mouse Model에서 BRD4 의존성 암세포 성장을 억제하는 것을 보고하였고, JQ1 및 그 유도체에 대한 Drug 역할 규명 연구의 중요성이 최근 주목받고 있는 상황이다. 이에 소개할 본 연구의 내용은 JQ1 유도체인 G2841819X Inhibitor에 대한 Target Engagement 연구이며, BRD4를 Target으로 하는 Diazirine 기반 신규 AfBP Chemical Tag 개발과 이를 활용한 질량 분석 Chemoprofiling를 통한 새로운 Off-Target 도출에 관한 내용을 설명하고자 한다.
연구결과
1. Minimalist Photocrosslinker 개발
연구팀의 이전 BRD4 Linker 개발을 통해 만들어진 Chemical Tag는 크게 2가지로 (1) Alkyne (2) Cyclopropene 버전 (각각 BD3, BD2)이 그것이다 (그림 2-A). 그림 2-A에서 보듯 BD3은 Azide기를 가진 Reporter와 Copper-catalyzed Azide-Alkyene Cyclo-addition(CuAAC) 반응을 하고, BD2는 in situ Bioimaging의 목적을 위해 Cyclopropene과 Tetrazine-Reporter와 Inverse Electron-Demand Diels-Alder (IED-DA) 반응이 일어날 수 있도록 하였다. 비록 화합물 처리 후에 세포 레벨에서 보이는 Non-Specific한 Background 문제가 있었지만 개발된 Linker는 세포 Imaging 과 질량분석을 동시에 할 수 있기 때문에 세포에서 관찰되는 형광 이미지 정보와 더불어 질량분석을 통한 Proteome Profiling 결과를 함께 비교 분석하여 Target 단백질 확인을 더 효과적으로 할 수 있다는 장점이 있었다 (5, 6). 이에 본 연구의 핵심은 BD Chemical Tag의 Dual Function에 착안해 BRD4 와 Inhibitor의 상호작용 연구를 위한 Next Generation Diazirine-Based Photocrosslinker를 새로이 합성하는 것에 있다 (그림 2-B).
새롭게 만들어진 Linker는 Click Handle로 Alkene(D8), Azide(N8), Tetrazine(T6)을 각각 포함하고 있으며 Terminal에 Primary Amine이 있어 BD Inhibitor(GW841819X)와 EDC Coupling을 통해 BD5,6,7 Photocrosslinker로 전환시켰다 (그림 2-B). 특히 Azide-Containing Linker(N8)은 One Pot 조건에서 Diaziridine-to-Diazirine Oxidation이 잘 발생되지 않았고 반응물로 Protected Diol 혹은 Azidation을 통한 Diazirine 합성은 잘 일어나지 않았다. Tetrazine-Containing Linker(T6)는 상대적으로 Harsh한 Diazirine 반응 조건으로 인해 Tetrazine이 대부분 Decompose되는 문제와 함께 Alkylation Specificity가 보이지 않아 다양한 Path를 통해 반응 조건을 최적화하였다. 이렇게 해서 친핵성도가 증가하고 Alkylation Specificity가 증가된 Tetrazine-Containing Secondary Amine(Z1)을 이용해 BD8을 합성할 수 있었다 (그림 2-B).
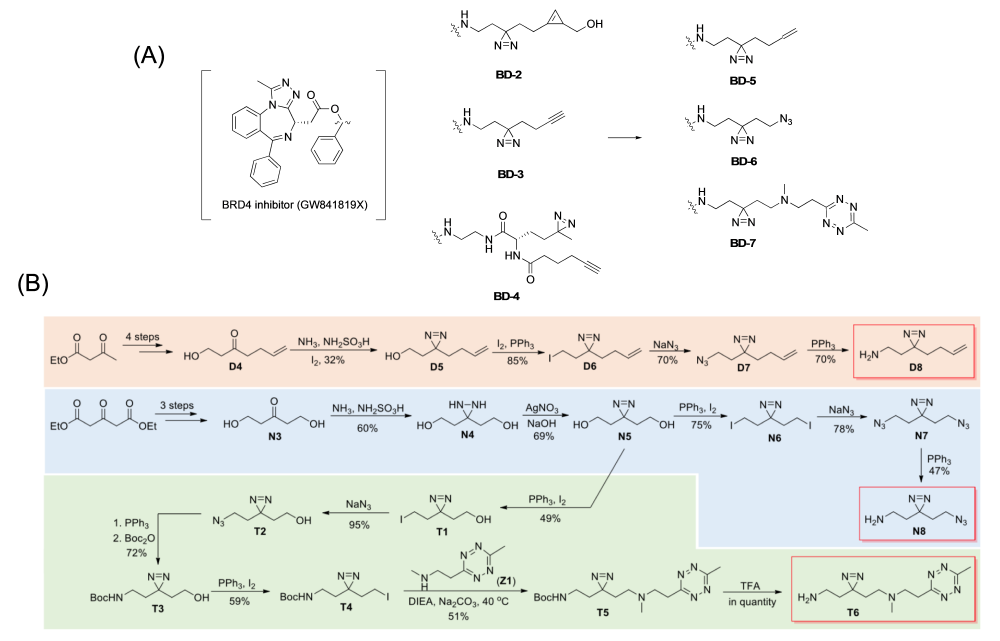
2. Minimalist Photocrosslinker Performance 분석
앞서 설명했듯 비록 동일한 Inhibitor Scaffold를 가지지만 Cell Imaging 및 MS Profiling을 위한 다양한 Click 작용기의 구조적 차이로 인해 BD Photocrosslinker 구조는 다르게 된다. 이에 Whole Proteome Level에서 BRD4 Inhibition, Live Cell Imaging, in situ Proteome Labeling Performance가 화합물 종류에 따라 어떠한 차이를 가지는지 분석하였다.
먼저 Single BRD4 단백질과 BD2~7을 in vitro 상에서 반응 후 분석하여 가장 적은 레이블링 효율을 보인 BD5를 제외한 나머지 화합물을 가지고 세포 내 Endogenous BRD4 및 BRD4 Downstream Protein에 대한 Inhibition 패턴을 분석하였다. BRD4 Inhibition 은 c-Myc Down-Regulation을 발생시키는 것으로 알려져 있다. 따라서 BD 화합물을 처리 시 Wild Type BRD4 Inhibitor(WT)와 동일하게 c-Myc 억제 신호를 보였다. 이와 같은 결과는 화합물과 BRD4 간의 세포 내 in situ 결합 후 BRD4(152kDa) 단백질만 선택적으로 Purification하여 화합물의 On-Target Interaction 효율 분석을 통해 더 확실하게 확인할 수 있었다.
두 번째로 Live Cell 이미징이 가능하도록 설계된 BD2,6,7의 효율을 확인하기 위해 Click Reaction Pair에 맞게 디자인된 Fluoro-Tetrazine(FL-TZ1), Fluoro-Dibenzocyclooctyne(FL-DBCO), Fluoro-Transcycloctene(FL-TCO)를 이용해 BRD4를 Target으로 Live-Cell Imaging을 하였다 (그림 3-A). BRD4는 세포 Transcription에 관여하는 단백질로 Nucleus에 존재하며 그림 3-C 1,3,5 패널에서 보듯 BD2,6,7은 서로 다른 Nucleus 염색 패턴을 보였다. 특히 7,8,9의 Anti-BRD4 항체를 이용한 Immunofluorescence(IF) Imaging 결과와 BD6/DBCO Pair를 이용한 Live Cell 이미징 패턴이 매우 유사하여 BD6이 가장 좋은 Imaging Performance를 가졌다.
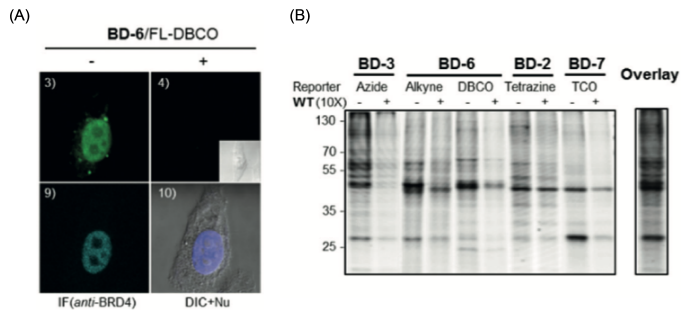
그림 3. (A) BD2,6,7 및 Pair-Reporter를 이용한 Live Cell Imaging 화합물 종류에 따라 서로 다른 형광 Morphology를 관찰할 수 있었다. (B) 화합물의 직접적인 S/N Ratio를 확인하기 위해 Click Chemistry를 통한 in situ Proteome 분석에 대한 결과로 BD3은 -/+ WT에서 동일 단백질 위치에 Turn-On/Off를 보여 Background가 가장 적은 것으로 판단되며 BD6/Alkyne 역시 비슷한 신호를 보여주고 있다. 반면 BD7의 경우에는 형광 시그널이 매우 적고 다른 Linker와는 다른 패턴으로 관찰이 되었다.
세 번째로 앞선 Inhibition 효율 측정과 다르게 화합물의 in situ Labeling 패턴을 직접적으로 보기 위해 BD Linker와 Click Pair 형광 Reporter를 이용한 Gel에서 형광 패턴을 분석하였다 (그림 3-B). 이러한 in situ Labeling이 이전의 BRD4 Antibody를 사용한 Inhibition 효율 측정 결과와 다른 점은 BD Tag의 S/N Ratio를 더 명확하게 알 수 있다는 점과 함께 이러한 Background 시그널 확인을 통해 MS 프로파일링에 적용 가능한 화합물을 선별할 수 있다는 점이다. 그림 3-D 에서 보듯 WT이 존재 시 모든 Linker는 시그널이 줄어드는 동일한 패턴을 보였지만 대부분 40-75kDa 사이에서 서로 다른 형광 시그널을 보였으며 실제 BRD4(152kDa)는 관찰되지 않았다. 이와 같은 결과는 앞서 설명한 BRD4(152kDa) Inhibition과는 상반되는 결과로 40-70kDa 사이가 Off-Target Region이 될 수 있다는 것을 가정할 수 있다. 특히 BD3 의 경우 WT Inhibitor 처리 시 동일 분자량 단백체들이 줄어들어 화합물 자체의 Non-Specific Background가 적은 것으로 판단할 수 있고, BD6-Alkyne Pair도 상대적으로 적은 Background를 보였으며, 그림에는 없지만 BD6/DBCO Pair는 반응 시간이 오래될수록 더 많은 Background 신호가 관찰되어 (8) DBCO Click Reaction 반응 시 2μM 이하의 농도와 30min 반응 시간이 Background로 인한 MS 분석 오차를 줄이는 것으로 확인하였다.
이처럼 in vitro 및 in situ 분석을 통해 동일한 BRD4 Inhibitor를 가지면서 다른 Click 작용기를 가진 화합물들에 대한 Performance 차이를 확인할 수 있었고, 특히 Live-Cell Imaging과 in situ Labeling을 동시에 하기 위해서는 Size, Stability, Bio-Orthgonality 측면에서 가장 좋은 특성을 가지는 Azide/BD6의 구조가 알맞은 것을 알 수 있었다.
3. 질량 분석 기반 Off-Target 도출
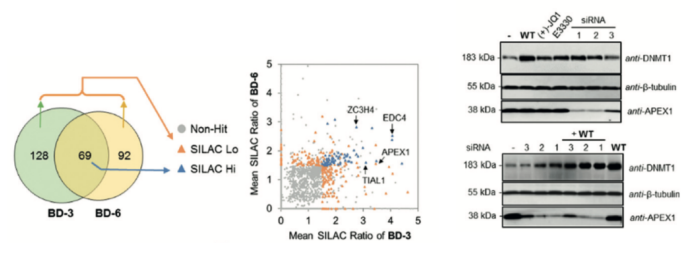
그림 4. SALIC 기반 Quantitative MS-Profiling을 이용한 JQ1 Off-Target 도출 방법. MS 신호를 기반으로 BD3/6에서 분석된 SALIC Low/High 단백체 프로파일링 한 결과를 토대로 siRNA를 이용한 APEX1 Inhibition 시 Down-Regulation 되는 DNMT1를 관찰할 수 있었다.
앞선 Performance 비교를 통해 도출된 BD3,6을 이용한 질량 분석 기반 단백체 프로파일링은 BD 화합물이 어떠한 단백체에 레이블링이 되는지를 확인할 수 있게 한다. 이를 위한 SILAC(Stable Isotope Labeling by Amino acids in Cell culture) 기반 Quantitative MS Profiling 기반 단백체 분석 방법은 동위원소로 치환된 Amino acid를 포함하는 Medium으로 세포를 배양하여 단백체의 Amino acid를 동위원소로 치환하게 하는 것으로써 이렇게 치환된 단백체는 +/- 화합물 조건 시 화합물로 인해 Down- or Up-Regulation 되는 세포 내 단백체 수 변화로 인해 Whole Proteome Leve에서 단백체의 질량 Ratio가 변하게 된다 (9). 이와 같은 원리로 BD 화합물을 처리한 세포를 SILAC 기반 MS 분석하여 (1) F/R 질량 Ratio>1.5 (2) Ratio>1.5 중 <0.67의 Coefficient of Variation을 가지는 단백체들을 검출했고 이중 BD3/6에서 모두 검출되는 High Ratio 단백체 69개와 각각에서 검출되는 220개들의 단백체를 프로파일링 하였다. 이렇게 프로파일링 된 단백체 중 BRD4와의 구조 유사성이 높고 Cellular Localization 분석을 통해 Nucleus에 존재하는 APEX1, TIAL1, EDC4, ZC3H4 4개 단백질로 Off-Target 후보군을 좁혔다. 특히 이중 APEX1 Inhibiton 시 DNMNT1의 Down-Regulation 되는 현상에 주목해 APEX1 Gene Knockdown을 통한 DNMT1 발현 분석으로 DNA-(Apurinic or Apyrimidinic Site) Lyase 단백질 APEX1이 신규 BRD4 Inhibitor의 Off-Target이라는 사실을 밝혔다 (그림 4).
이번 연구는 in situ Drug Profiling에 사용 가능한 새로운 Alkyl Diazirine Based AfBP 화합물을 사용해 서로 다른 종류의 Chemical Tag가 어떻게 화합물의 Performance에 영향을 미치는지 근거를 명확히 설명하여 신규 Chemical Tag 합성 시 다양한 종류의 Bioorthgonal Tags의 비교 분석이 필요함을 제시하였고, 새로이 합성된 화합물을 바탕으로 고분해성 질량분석을 통한 BRD4 Inhibitor Off-Target인 APEX1을 도출하였다는 점에서 큰 의미가 있다.
참고문헌
1. Chan, E. W., Chattopadhaya, S., Panicker, R. C., Huang, X., Yao, S. Q. (2004) Developing photoactive affinity probes for proteomic profiling: hydroxamate-based probes for metalloproteases. J Am Chem Soc 126, 14435-14446.
2. Mackinnon, A. L., Taunton, J. (2009) Target Identification by Diazirine Photo-Cross-linking and Click Chemistry. Curr Protoc Chem Biol 1, 55-73.
3. Kleiner, P., Heydenreuter, W., Stahl, M., Korotkov, V. S., Sieber, S. A. (2017) A Whole Proteome Inventory of Background Photocrosslinker Binding. Angew Chem Int Ed Engl 56, 1396-1401.
4. Filippakopoulos, P., Qi, J., Picaud, S., Shen, Y., Smith, W. B., Fedorov, O., Morse, E. M., Keates, T., Hickman, T. T., Felletar, I., et al. (2010) Selective inhibition of BET bromodomains. Nature 468, 1067-1073.
5. Patterson, D. M., Nazarova, L. A., Prescher, J. A. (2014) Finding the right (bioorthogonal) chemistry. ACS Chem Biol 9, 592-605.
6. Li, Z., Wang, D., Li, L., Pan, S., Na, Z., Tan, C. Y., Yao, S. Q. (2014) "Minimalist" cyclopropene-containing photo-cross-linkers suitable for live-cell imaging and affinity-based protein labeling. J Am Chem Soc 136, 9990-9998.
7. Dubinsky, L., Jarosz, L. M., Amara, N., Krief, P., Kravchenko, V. V., Krom, B. P., Meijler, M. M. (2009) Synthesis and validation of a probe to identify quorum sensing receptors. Chemical Communications 2009, 7378-7380.
8. Laughlin, S. T., Baskin, J. M., Amacher, S. L., Bertozzi, C. R. (2008) In vivo imaging of membrane-associated glycans in developing zebrafish. Science 320, 664-667.
9. Ong, S. E., Mann, M. (2006) A practical recipe for stable isotope labeling by amino acids in cell culture (SILAC). Nat Protoc 1, 2650-2660.
첨부파일