
생화학분자생물학회입니다.
생체 내 뉴클레오솜 배열 상태 조절을 통한 후성 유전학적 유전자 전사 조절
작성자
관리자작성일자
2015-12-01조회수
1679생체 내 뉴클레오솜 배열 상태 조절을 통한 후성 유전학적 유전자 전사 조절
Epigenetic Regulation of Transcription through Nucleosome Positioning in vivo

이준우 |

이대엽 |
1. 서론
지구상의 모든 생물은 기본적으로 핵산의 염기 서열을 통하여 기본적인 생물학적 정보를 ‘게놈(Genome)’이라는 유전학적인 정보로써 보관하고, 사용하며, 후대에 물려준다. 생물은 진화해 감에 따라 자신의 유전 정보를 점차적으로 늘려갔고, 이에 진핵 생물은 자기 자신에 대한 방대한 정보를 담고 있는 이 게놈(Genome)을 핵(Nucleus)이라는 제한된 공간에 효율적으로 담기 위해 자신의 게놈을 뉴클레오솜(Nucleosome)이라는 단위로 이루어진 염색질(Chromatin)이라는 응축된 구조로 보관하게 된다. 이 염색질 구조는 결과적으로 그 안에 응축되어있는 DNA를 주형(Template)으로 사용하여 일어나는 다양한 생물학적 현상들 – DNA 복제(Replication), DNA 손상 복구(DNA Damage Repair) 및 유전자 전사(Transcription) – 이 일어나는 것을 방해하는 물리적 장벽으로 작용하게 된다. 따라서, 이같이 감추어진 유전 정보들을 사용하기 위해서는 그 응축된 뉴클레오솜과 염색질 구조를 얼마나 효율적으로 풀어주고, 다시 원래대로 돌려놓는가에 달려있다. 이에 더하여, 최근 발전한 차세대 염기서열 분석기술을 기반으로 한 연구들에서 이렇게 형성된 염색질 구조가 게놈 DNA를 주형으로 사용하는 생물학적 현상에 대한 방해자의 역할만 하는 것이 아니라 여러 염색질 관련 단백질들(Chromatin-Associated Proteins)이 알맞은 위치에 와서 작용할 수 있도록 돕는 역할을 수행한다는 사실이 밝혀지고 있다. 그렇기에 생체 내 뉴클레오솜 배열의 상태를 조절하는 분자 기전이 어떻게 후성 유전학적으로 유전자 전사를 조절하는가를 밝혀내는 일이 오늘날 염색질 생물학(Chromatin Biology)에서 다루는 중요한 사안이 된 것이다.
이에 본 논단에서는 우선 게놈 응축의 최소 단위인 뉴클레오솜과 그것들이 모여서 만들어지는 염색질에 대하여 알아본 후, 이 뉴클레오솜과 염색질 구조가 유전자 발현에 미치는 영향을 알아보고자 한다. 그 후 생물이 어떻게 이러한 뉴클레오솜 및 염색질 구조 안에 감추어져 있는 유전자 발현을 후성 유전학적으로 조절하는지에 대해 알아보며, 마지막에는 이 염색질과 후성 유전학적 유전자 조절 사이의 관계에서 풀어야 할 질문들에 대하여 다루어 보고자 한다.
2. 본론
2.1 뉴클레오솜의 구조 및 뉴클레오솜 배열의 특징과 그에 따른 역할들
뉴클레오솜은 약 147bp의 DNA가 2개의 H2A-H2B 이량체와 1개의 H3-H4 사량체로 구성된 히스톤(Histone)이라고 하는 팔량체를 약 1.7번 감으면서 생기는 구조이다. 이와 같이 뉴클레오솜에 결합하는 DNA는 자신의 유연성(Flexibility)를 충분히 보장하여 히스톤 팔량체에 감길 수 있도록 하는 염기 서열을 가지고 있어야 하는데, 이에 관여하는 것이 바로 상대적으로 염기의 크기가 작아 DNA의 유연성을 증가시켜줄 수 있는 아데닌(Adenine)과 티민(Thiamine)이 중심이 되는 A-T 풍부 서열(A-T Rich Sequence)이다. 이렇게 만들어지는 뉴클레오솜은 서론에서도 언급한 바, 일차적으로는 방대한 분량의 게놈을 효율적으로 보관할 수 있도록 해주는 압축 역할을 함과 동시에 그 뉴클레오솜에 감겨있는 DNA 염기 서열이 다른 물질들 – 전사 인자(Transcription Factor)와 같이 DNA에 붙거나, RNA 중합 효소 II(RNA Polymerase II)와 같이 DNA를 형판(Template)로 사용하거나, 뉴클레아제(Nuclease)와 같이 DNA를 기질(Substrate)로 사용하는 단백질 등 – 과 반응하는 것을 막는 역할을 한다. 재미있는 사실은, 이렇게 히스톤에 감겨 있는 동일한 염기 서열의 DNA라도 뉴클레오솜의 중심(Dyad)으로부터 DNA가 멀어지면 멀어질수록 가역적으로 그 DNA와 히스톤 사이의 국지적인 결합이 떨어지는 DNA 호흡(DNA Breathing)이 일어나는 빈도가 현저하게 차이가 나며, DNA의 두 홈 – Major/Minor Grooves – 중 어느 쪽이 히스톤과 붙어 있느냐에 따라서도 그 뉴클레오솜의 특성에 변화가 나타나게 된다. 이 뿐만 아니라, 뉴클레오솜 내에 히스톤 H4의 꼬리(Tail)는 서로 이차원적으로나 삼차원적으로 가까이 있는 다른 뉴클레오솜의 H2A-H2B 이량체와 상호 작용을 하여 뉴클레오솜 사이의 응축(Inter-Nucleosomal Packing)을 유도하여, 보다 복잡한 염색질 구조를 이루는 것을 촉진한다고 알려져 있다.
이러한 뉴클레오솜은 게놈 상에서 서로 짧게는 몇 십 염기 쌍(bp; Base Pairs)에서 길게는 몇 백 bp씩 떨어져 있는 뉴클레오솜 배열(Nucleosome Array)을 이루고 있는데, 이렇게 뉴클레오솜들 사이에서 히스톤 단백질에 감겨져 있지 않은 DNA를 연결자 DNA(Linker DNA)라고 부른다. 이러한 뉴클레오솜 배열이 이루는 염색질의 구조와 그 구조의 의미에 대한 연구는 최근 십 수년 간 눈부시게 발전되어 온 차세대 서열분석법(NGS; Next-Generation Sequencing)을 기반으로 한 염색질 면역 침강 서열분석법(ChIP-seq; Chromatin Immuno-Precipitation Sequencing)과 각종 RNA에 대한 시퀀싱(mRNA-seq, Total RNA-seq, Small RNA-seq 등), 그리고 MNase를 이용한 단일 뉴클레오솜 서열분석법(MNase-seq or Mono-Nucleosome Sequencing; 그림 1 (A)) 등을 통해 활발히 이루어져 왔으며, 다음과 같은 사실들을 밝혀내었다.
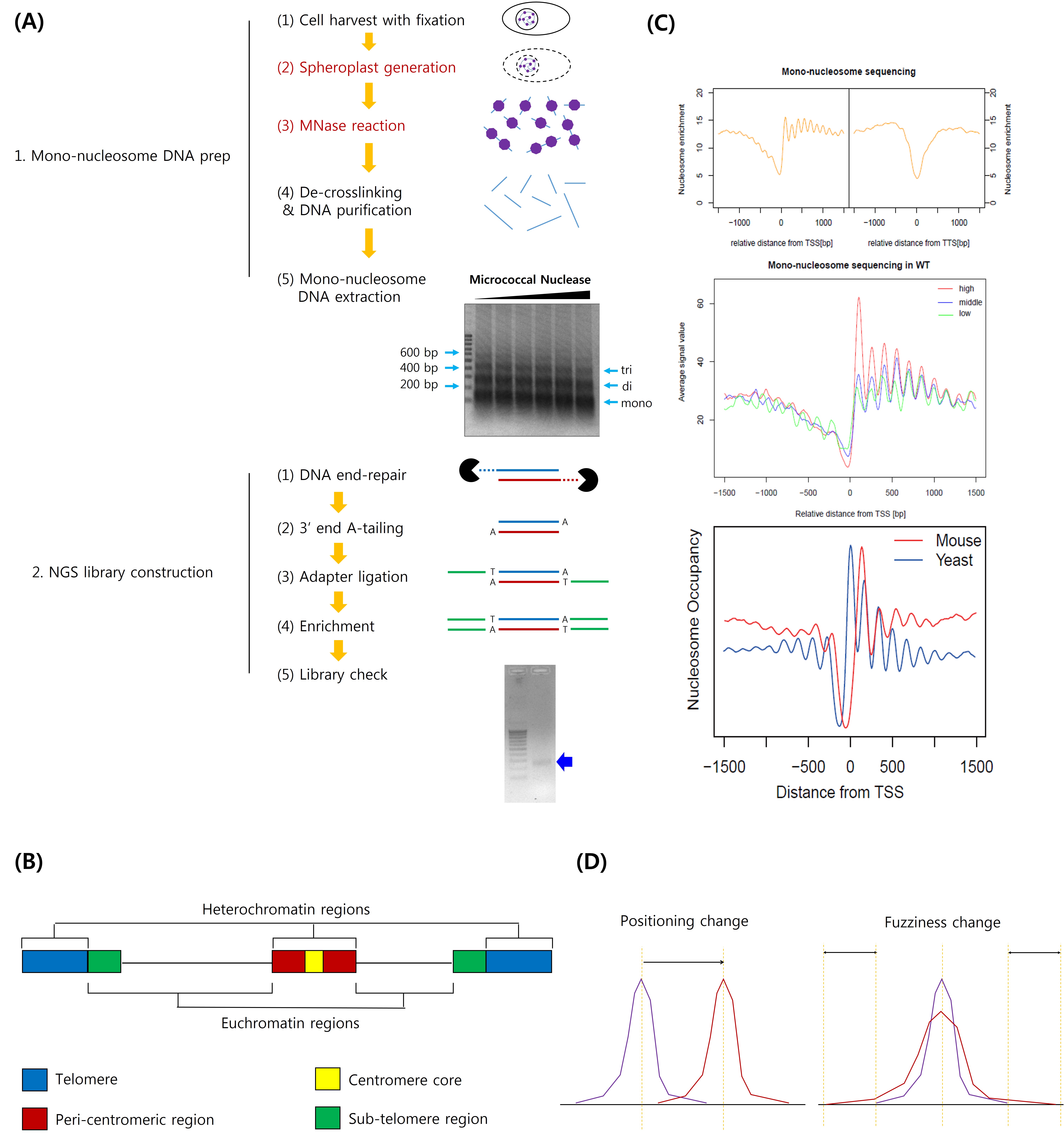
그림 1. 염색질 구조와 뉴클레오솜 시퀀싱에 대한 개요
(A) 뉴클레오솜 시퀀싱에 대한 모식도. 붉은색 처리된 단계들이 실험의 성공 여부를 결정하는 중요한 단계들이다. (B) 효모 염색질의 구조에 대한 모식도. 크게 진정 염색질과 이질 염색질로 나누어 진다. (C)뉴클레오솜 위치 선정에 대한 특징들. 전사 시작 위치 및 전사 종결 위치 쪽에 특이적인 뉴클레오솜 자유 구간이 존재하고 (위), 유전자 전사율이 높을수록 뉴클레오솜 위치 지정이 더욱 선명하며 (중간), 상대적으로 전사 시작 위치보다 먼 위치에서 +1 뉴클레오솜이 형성되는 쥐의 경우, 효모에서보다 +1 이후의 뉴클레오솜 위치 지정이 더 불확실하다. (D) 뉴클레오솜 배열 및 사용량 변화는 크게 위치와 흐릿함의 변화로 나타난다.
(1) 염색질은 상대적으로 심하게 응축되어 있는 이질 염색질(Heterochromatin)과 상대적으로 적게 응축되어 있는 진정 염색질(Euchromatin)로 나누어 진다 (그림 1 (B)). 진핵 생물의 염색질은 기본적으로 핵 안에 안정적으로 존재하기 위해 더 응축된 상태인 이질 염색질을 기본 상태로 삼으며, 주형으로 사용할 필요가 있는 DNA 부근을 다양한 기작들을 통해 진정 염색질로 바꿈으로써 그 주형 DNA를 이용하고자 하는 물질의 접근을 물리적으로 허용하게 된다. 또한, 염색질의 양 끝에 존재하는 말단 소립(Telomere)과 가운데에 존재하는 동원체(Centromere)쪽에는 안정적인 이질 염색질이 존재하여 비정상적인 전사가 일어나는 것을 방지하고 다양한 생물학적 현상에 이 구조들을 이용하고 있다.
(2) 진정 염색질 위치 중, 염색질 관련 단백질들이 많이 오는 위치에는 뉴클레오솜이 특이적으로 적은 뉴클레오솜 자유 구간들(NFR; Nucleosome Free Region. NDR; Nucleosome Depleted Region 라고도 부른다)이 존재하여 그 단백질들의 위치 지정(Targeting)에 중요한 역할을 한다 (그림 1(C)). 이러한 위치들 중 가장 대표적인 곳들은 유전자를 기준으로 전사 시작 위치(TSS; Transcription Start Site)와 전사 종결 위치(TTS; Transcription Termination Site), 그리고 인핸서(Enhancer) 근처에 각각 존재하는 뉴클레오솜 자유 구간들이다. 이 위치들 중, 전사 시작 위치의 상류(Upstream)쪽에 위치하는 뉴클레오솜 자유 구간은 대체로 다양한 전사 인자들이나 전사 전 복합체(PIC; Pre-Initiation Complex)를 이루는 요소들이 붙는 프로모터(Promoter) 구간과 일치한다. 이곳과 인핸서 위치에서는 뉴클레오솜 자유 구간이 인핸서에 오는 단백질들의 위치 지정 및 인핸서의 활성에 중요한 역할을 수행한다고 알려져 있다. 이에 반해, 전사 종결 위치 하류(Downstream)에 위치하는 뉴클레오솜 자유 구간에 대해서는 아직까지 많은 연구들이 진행되지 않았기에, 이 위치의 뉴클레오솜 자유 구간이 발생하는 기작과 그 역할에 대하여 많은 연구가 요구되고 있는 실정이다.
(3) 진정 염색질 위치 중 뉴클레오솜이 가장 잘 배열되어 있는 곳은 유전자 발현이 일어나는 개방형 해독틀(ORF) 위치이다 (그림 1 (C)). 이 위치에서 위치 지정이 가장 선명한 곳은 바로 전사 시작 위치의 하류에서 가장 가까운 곳에서 나타나는 +1 뉴클레오솜인데 이 뉴클레오솜은 프로모터와 전사 시작 위치에서 가까울수록, 또 그 유전자의 전사율이 높을수록 선명하게 나타나는 특징을 보인다. 또한 이 뉴클레오솜이 위치 지정이 분명한 곳일수록, 그리고 그 유전자의 길이가 상대적으로 짧을수록 그 하류에서 규칙적으로 나타나는 뉴클레오솜 배열들 또한 선명한데 이러한 점으로부터 +1 뉴클레오솜이 그 하류 뉴클레오솜들의 위치 지정의 기준으로써 작용하고 있다고 생각되고 있다. 이러한 구조적 특징은 효모와 포유류를 비교할 때 더욱 분명한데, 효모는 포유류에 비교하여 상대적으로 프로모터와 +1 뉴클레오솜 간격이 짧고 그 유전자의 길이 또한 짧기에 이러한 뉴클레오솜 배열이 더욱 선명하다는 특징이 있다. 이처럼 잘 배열된 뉴클레오솜들은 프로모터에서 이루어지는 일련의 과정들을 조절하거나 유전자 전사 과정에서 뉴클레오솜 장벽(Nucleosome Barrier)으로 작용하여 RNA 전사를 수행하는 RNA 중합효소의 반응 진행성(Processivity)에 영향을 줄 수 있기에 생물은 이 뉴클레오솜들을 역동적으로 움직일 필요가 있다. 또한, 이렇게 잘 이루어져있는 뉴클레오솜 배열은 유전자 내부에서 의도치 않은 비정상적인 전사물(Cryptic Transcript)이 발생하여 생물의 전사체(Transcriptome)가 무너지는 것을 막는다.
(4) 뉴클레오솜의 사용량(Occupancy)은 크게 위치 지정(Nucleosome Positioning)과 흐릿함(Fuzziness)으로 결정된다 (그림 1 (D)). 뉴클레오솜의 사용량이라는 것은 뉴클레오솜이 ‘특정 부근에 얼마나 있는가’를 표현하는 일종의 면적을 나타내는 말이다. 이 때, 그 뉴클레오솜이 가장 많이 존재하는 위치가 다른 특정 위치 – 대표적으로 전사 시작 위치나 야생형 세포(Wild Type Cell)에서 나타나는 뉴클레오솜의 위치 – 를 기준으로 하여 볼 때 얼마나 일정한가를 살펴볼 때 쓰는 용어가 바로 위치 지정이며, 그 뉴클레오솜이 그 주변의 뉴클레오솜과 얼마나 잘 구분되어 보이는가를 나타낼 때 사용되는 것이 바로 그 뉴클레오솜의 흐릿한 정도이다. 따라서, 특정 돌연변이 상황에서 어떤 요소에 어떠한 형태로 변화가 나타나는가를 관찰함으로써 그 돌연변이가 일어나는 유전자의 야생형 결과물이 뉴클레오솜 배열에 미치는 영향을 유추해 낼 수 있다. 대표적인 예시로 특정 단백질이 뉴클레오솜들 사이의 간격을 조절하는 경우(Nucleosome Spacing), 뉴클레오솜들의 위치들이 이동하게 되며, 뉴클레오솜의 특징이 변하는 경우 – 뉴클레오솜의 번역 후 변형 (PTM; Post-Translational Modification) 상태가 변하거나 이형 히스톤(Histone Variant)이 기존 히스톤을 대체하여 뉴클레오솜에 들어가게 되는 경우 등 – 흐릿함에 변화가 일어날 가능성이 높다.
2.2 뉴클레오솜 위치 선정의 분자 기전
초창기 연구에서 뉴클레오솜 배열이 특정 DNA 염기 서열들에서 열역학적으로 선호된다는 사실이 드러남에 따라 염색질의 구조가 유전체의 서열에 의해 예정되어 있을 수 있다는 가능성이 제시되었었다. 하지만 시간이 흐름에 따라 전사 인자에서부터 RNA 중합효소, 그리고 ATP-의존성 크로마틴 리모델러(ATP-Dependent Chromatin Remodeler) 등에 이르는 후성 유전 단백질들 또한 뉴클레오솜 위치 선정에 지대한 영향을 미친다는 사실이 밝혀졌다. 이에, 본 글에서는 지금까지 알려진 뉴클레오솜 위치 선정 요소들을 살펴보고자 한다.
(1) DNA 염기서열: 기본적으로 단일 히스톤은 DNA 서열에 특이적 결합능을 가지지 못하지만 히스톤 단백질 사이의 결합능이 DNA의 Minor Groove와 상호작용하는데 중요하다고 알려져 있다. 초기 연구들에서 성게의 5S 리보솜 RNA(Sea Urchin 5S Ribosomal RNA (rRNA))의 DNA 서열과 같이 히스톤 팔량체가 선호하는 DNA 염기서열이 존재한다는 사실이 알려졌고, Drew & Travers의 연구에서 시험관에서(in vitro) 생성된 뉴클레오솜이 DNA에 대해서 회전적 선호(Rotational Preference; AT 디뉴클레오티드(Dinucleotide)의 주기성)를 보인다는 것을 알게 되었다. 이 연구에 따르면, 시험관에서 만들어진 뉴클레오솜 DNA는 소홈(Minor Groove)이 뉴클레오솜 중심(Nucleosome Core) 내부 방향으로 향해 있는 AT 디뉴클레오티드가 일반적이며 대략 10.15bp 주기의 디뉴클레오티드와 트리뉴클레오티드(Trinucleotide)를 가진다. 하지만 AT 디뉴클레오티드 주기를 기반으로 한 뉴클레오솜 예측 모델들은 프로모터 뉴클레오솜 결실(Promoter Nucleosome Depletion)과 같은 생체 내에서 나타나는 특이적인 뉴클레오솜 위치 선정을 예측하는 데 실패했다. 하지만 다양한 실험을 통해 뉴클레오솜 위치 선정에 배제되는 DNA 서열(예를 들어 Poly(A))의 존재도 알려지게 되었다. 결론적으로, DNA 서열은 열역학적인 개념에서 뉴클레오솜 위치선정에 중요하지만 이것만으로는 충분하지 않다.
(2) 뉴클레오솜 자유 구간 주변에 위치한 뉴클레오솜들: 미국 스탠포드 대학교의 Kornberg 교수와 Stryer 교수의 통계적 위치 지정(Statistical Positioning) 가설에서 기원한 이론과 실제 세포 내 뉴클레오솜 위치 선정 결과에 따르면, 뉴클레오솜이 특정 위치에 대해 본질적인 선호도를 가지고 있지 않다고 하더라도 뉴클레오솜이 주어진 DNA의 끝부분에 고정되어 있다면, 그 이후에 들어오는 뉴클레오솜의 위치는 한정적일 수밖에 없다. 이를 바탕으로 실제 세포 내에서는 매우 강하게 위치를 선점한 뉴클레오솜을 기준으로 하여 주변의 뉴클레오솜 위치가 확률적으로 결정될 수 있으며, 그 뉴클레오솜들 사이의 간격을 조절하여 규칙적인 배열을 만드는 데에 크로마틴 리모델러가 필요하다고 생각되고 있다. 이러한 사실을 가장 잘 반영하는 곳이 바로 프로모터 부위의 +1과 -1 뉴클레오솜들이다. 앞에서도 설명한 바, 이 뉴클레오솜들은 프로모터와 전사 시작 위치에서 가까울수록 그 유전자의 전사율이 높을수록 그 위치가 분명하고 선명하며, 이 뉴클레오솜이 위치 지정이 분명한 곳 일수록 그리고 그 유전자의 길이가 상대적으로 짧을수록 그 하류에서 규칙적으로 나타나는 뉴클레오솜 배열들 또한 선명하다.
(3) 전사 인자: 효모에서 유전자의 프로모터들은 시험관에서보다 생체 내(in vivo)에서 훨씬 더 큰 뉴클레오솜 결실을 보이는데, 이는 뉴클레오솜 자유 구간이 이곳에 붙어서 역할을 수행하는 다양한 전사 인자들 – 보편적/특정 전사 인자(General/Specific Transcriptional Factor)나 전사 전 복합체 – 에 의해서 좌우될 수 있음을 암시하고 있었다. 따라서 전사 인자와 히스톤이 DNA에 붙기 위해 경쟁할 것이라고 추정되어 왔는데, 이를 Workman & Kingston이 20여 년 전에 GAL4 전사 인자의 뉴클레오솜 결합이 히스톤 샤페론(Histone Chaperone)의 존재 하에 뉴클레오솜 DNA에서 히스톤을 떼어낼 수 있음을 보임으로써 증명하였다. 이러한 결과는 DNA 결합 단백질들이 생물 내에서 뉴클레오솜 위치 선정에 중요한 역할을 수행할 수 있음을 암시하며 이는 앞서 언급한 고등 동물의 인핸서 및 프로모터에 붙은 인핸서들과 이 위치들에서 나타나는 뉴클레오솜 결실을 설명하여 준다.
(4) 크로마틴 리모델러와 히스톤 샤페론: 앞에서도 설명한 것처럼 DNA 염기서열 자체만으로는 프로모터나 전사 종결 위치 부근, 그리고 인핸서상에 존재하는 뉴클레오솜 자유 구간을 완벽하게 재현해 낼 수 없고 설사 그 구간이 전사 인자들을 통하여 만들어진다고 하여도 그 구간들을 기준으로 나타나는 정렬되는 뉴클레오솜들은 확률적으로 분포되어질 뿐, 각각의 안정적인 위치를 스스로 지정할 수는 없다. 따라서 앞서 언급한 뉴클레오솜 자유 위치에 결합 가능한 전사 인자들이나 그 곳의 DNA에 크로마틴 리모델러들이 ATP에 내포되어 있는 인산 결합 에너지를 이용하여 뉴클레오솜 배열을 재정비 하는 과정을 수행하게 된다. 이 크로마틴 리모델러들은 크게 SWI/SNF, ISWI, INO80와 CHD의 네 가지 군으로 나누어지며 이들 중 ISWI와 CHD계열이 통상적인 뉴클레오솜 위치 지정에 가장 중요하며, 뉴클레오솜들을 안정화 하여 본래 유전자들의 전사체가 아닌 비정상적인 전사체가 발현되는 것을 막는다. 한편, SWI/SNF는 뉴클레오솜 축출(Nucleosome Eviction)에 기본적인 역할을 하고 INO80 계열 – INO80 복합체와 SWR 복합체 – 은 이형 히스톤 H2A.Z를 뉴클레오솜에 넣고 빼는 역할을 수행한다. 이와 다르게, 히스톤 샤페론은 ATP를 이용하지 않고 다양한 히스톤들 및 뉴클레오솜에 붙는 친연성(Histone/Nucleosome Binding Affinity)를 이용하여 염색질에 존재하는 뉴클레오솜의 안정성(Stability)에 영향을 주거나, 염색질 주변에 떠다니는 히스톤들을 붙잡아두는 완충 효과(Buffering Effect)를 수행하게 된다.
(5) RNA 중합효소: RNA 전사 기작은 전사가 일어나는 위치들의 크로마틴 구조에 상호적으로 영향을 미친다. 뉴클레오솜 위치 선정이 전사 프로그램에 미치는 영향을 밝히는 일은 효모를 이용한 Hörz 박사와 그 동료들의 PHO5 Promoter에 관한 연구에서 시작되어, 많은 연구자들이 탄소원 변화(Carbon Source Shift), 스트레스 반응(Stress Response), 그리고 교배 상황(Meiosis)에서의 뉴클레오솜 위치 선정과 사용량의 변화를 관찰했다. 이에 더하여, 초파리를 이용한 고온 스트레스에 대한 반응 및 인간의 CD4+ T 세포에서 T 세포 수용체(TCR; T Cell Receptor)의 활성을 조사하는 등의 연구가 수행되었다. 재미있는 점은, 시험관 상에서는 뉴클레오솜 배열이 유전자 전사에 물리적인 장애물로 작용하지만 생체 내에서는 그 유전자의 전사율이 높을수록 그 유전자의 프로모터 근처나 개방형 해독틀에서 뉴클레오솜 사용량이 높고 그 위치 지정이 분명하다는 점이다. 또한 특정 상황이나 조건에서 발현이 일어나거나 억제되는 유전자의 경우, 그 유전자 발현 상태에 따라 프로모터 부근의 뉴클레오솜들의 위치 및 사용량을 조절함으로써 그 유전자의 전사에 영향을 미친다고 알려져 있다.
2.3 뉴클레오솜 위치 선정 조절 기작과 후성 유전학적 유전자 전사 조절
앞에서도 언급한 것처럼 유전자 발현을 연구하기 위하여 수행된 RNA 중합효소 II에 의해 일어나는 전사에 대한 시험관 실험들은 이 RNA 중합효소의 반응 진행성이 뉴클레오솜이 없는 DNA 상태에서 뉴클레오솜이 있는 상태보다 훨씬 빠르게 나타나며, 뉴클레오솜이 유전자 전사에 대한 뉴클레오솜 방어벽(Nucleosome Barrier)로 작용함을 시사해 왔다. 그러나 놀랍게도, 생체 내에서는 뉴클레오솜들의 위치 선정이 상대적으로 뚜렷한 유전자들이 오히려 전사율이 높고 RNA 중합효소의 반응 진행성 또한 시험관 내 실험에서 뉴클레오솜이 없는 DNA 상태에서 일어나는 유전자 발현과와 일치한다. 이는 생체 내에서는 전사 중인 RNA 중합효소가 다가올 때는 그 뉴클레오솜 방어벽을 무너뜨렸다가 RNA 중합효소가 지나간 후에는 다시 그 뉴클레오솜을 원위치 시키는 기작이 효율적으로 일어나고 있음을 시사한다. 여기에서는 뉴클레오솜 위치 선정에 관여하는 후성 유전학적 요소들이 어떻게 유전자의 전사 기작을 조절하는지 각 요소 별로 살펴보도록 한다.
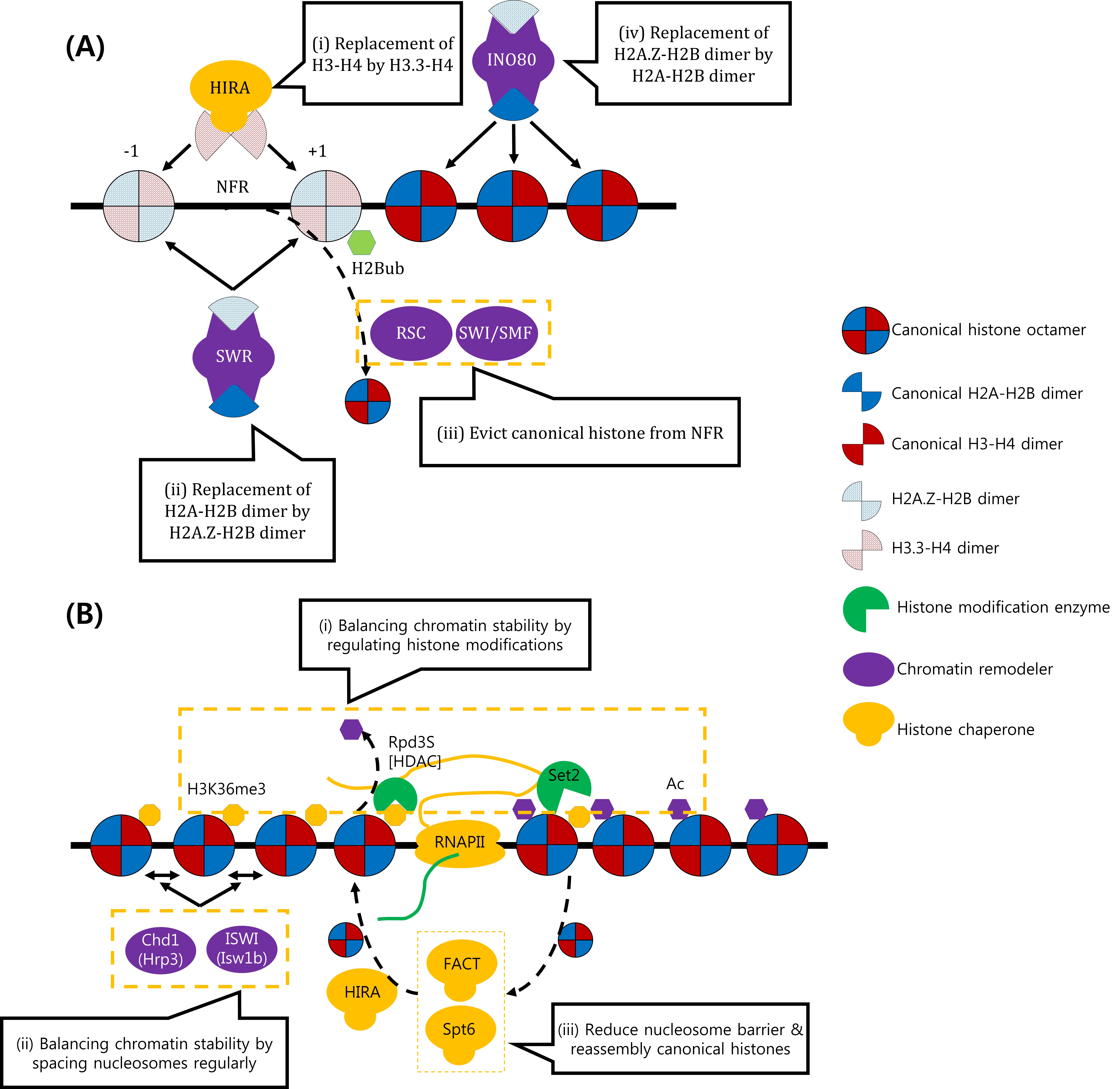
그림 2. 유전자 전사 상황과 뉴클레오솜 위치 지정 및 배열의 상관 관계
(A) 전사 시작 위치에서 후성 유전학적 뉴클레오솜 위치 및 배열 조절의 역할들. 주로 이형 히스톤 H2A.Z와 H3.3을 통해 유전자 전사 과정 조절을 돕는다. (B) 유전자 전사 신장 과정과 후성 유전학적 뉴클레오솜 위치 및 배열 조절의 역할들. 크게 염색질의 안정성 조절과 유전자 전사 상의 뉴클레오솜 방어벽 조절과 연관이 있다.
(1) 뉴클레오솜의 번역 후 변경 및 이형 히스톤들: 뉴클레오솜의 번역 후 변경은 크게 아세틸화(Acetylation), 메틸화(Methylation), 인산화(Phosphorylation), 유비퀴틴화(Ubiquitylation) 및 수모화(Sumoylation)로 나누어지며 히스톤의 다양한 위치들 중 특히 질소 말단에 존재하는 히스톤 꼬리 부근에서 가장 많이 일어난다. 현재까지 유전자 발현과 관련된 뉴클레오솜의 변역 후 변경은 H3와 H4에서 일어나는 아세틸화, H3K4, H3K36, H3K79에서 일어나는 메틸화, 그리고 H2B의 탄소 말단 꼬리에서 일어나는 단일유비퀴틴화 정도로 나누어 볼 수 있다. 이들은 종합적으로 뉴클레오솜 방어벽을 낮추거나 비정상적인 전사가 일어나지 않도록 염색질의 안정성을 유지시키는 역할을 하며 유전자 전사의 시작 및 신장 과정에서 RNA 중합효소의 진행을 돕는다 (그림 2). 유전자 발현에 가장 많이 영향을 미치는 이형 히스톤은 크게 H3.3와 H2A.Z로 볼 수 있다 (그림 2 (A)). 이 중 H3.3는 통상적인 H3와 단 4~5개의 아미노산만 차이가 나는데 이 작은 차이가 뉴클레오솜 사이의 상호 연락을 방해하는 역할을 하여 염색질의 전반적인 구조에 영향을 주게 된다고 알려져 있다. 또한 통상적인 H3는 S기에서만 발현되어 히스톤 샤페론 중 하나인 CAF1에 붙어서 DNA 복제 과정 동안 염색질에 들어간다고 알려져 있으나, H3.3는 세포 주기 동안 항상 발현되어 또 다른 히스톤 샤페론인 HIRA/DAXX/ATRX 붙어서 DNA 복제 과정과 무관하게 진정 염색질과 이질 염색질에 들어갈 수 있다. 그리고 H2A.Z는 세포 주기 동안 항상 발현되며 H2A.Z에 특화된 크로마틴 리모델러인 SWR 복합체에 의하여 특정 환경에 발현이 일어나는 유도 유전자들의 프로모터에 위치하여 H3.3와 함께 히스톤 팔량체를 이루는 경우, 일반적인 뉴클레오솜들에 비해 더욱 불안정한 상태가 되어 RNA 중합효소의 진행을 덜 방해할 것으로 생각되고 있다.
(2) 히스톤 샤페론과 크로마틴 리모델러: 앞에서 논한 바, 일부 히스톤 샤페론들과 크로마틴 리모델러들은 유전자 전사에 관여하는 H3.3와 H2A.Z를 기존에 존재하는 통상적인 히스톤들을 빼고 대신 넣기 위하여 함께 작용하고 있다. 이와 다르게, 유전자 전사 과정에서 통상적인 뉴클레오솜의 안정성과 위치를 조정하는 히스톤 샤페론들과 염색질 개조들도 존재한다. 히스톤 샤페론들 중 HIRA가 전사 시작 상황에서 +1 뉴클레오솜을 조절하고 (그림 2 (A)), 전사 신장 상황에서 Spt6, FACT 및 HIRA가 유전자 전사 지역에서 뉴클레오솜 방어벽을 낮추거나 전반적인 뉴클레오솜 사용량을 조절하는데 관여하고 있다 (그림 2 (B)). 이들 중에서도 특히 시험관 실험 및 생체 내 관찰을 통한 실험들에서 가장 잘 연구가 되어 있는 FACT는 유전자 전사 상황에서 RNA 중합효소와 직접 붙어서 전사가 많이 일어나는 프로모터 및 전사 지역에서 전사 전 복합체의 형성 및 전사 신장 복합체로의 변화, 그리고 전사 신장을 수행 중인 RNA 중합효소의 반응 진행성 모두에 관여한다고 알려져 있다. 한가지 재미있는 점은, 유전자 전사율에 비례하여 RNA 중합효소를 따라 유전자의 전사 지역으로 이동하는 FACT에 문제가 생기면 상대적으로 FACT의 사용량이 낮은 전사율이 낮은 유전자들과 이질 염색체 부근에서 비정상적으로 전사가 증가한다는 점인데, 이는 전사율이 높은 유전자에서의 RNA 중합효소의 반응 진행성 및 사용량이 감소하면 그 중합효소들이 다른 유전자들로 비정상적으로 이동할 가능성이 있음을 시사한다. 크로마틴 리모델러는 프로모터에서는 SWR와 RSC, 그리고 SWI/SNF가 중심이 되어 프로모터 부근의 뉴클레오솜 자유 구간의 구조 및 전사 전 복합체를 조절하는데 관여하고 있고 (그림 2 (A)) 전사 지역에서는 INO80가 H2A.Z-H2B 이량체가 이 위치의 뉴클레오솜에 들어가는 것을 막고 있으며 (그림 2 (A)), ISWI와 CHD가 뉴클레오솜들의 간격을 조절하여 비정상적인 전사가 일어나는 것을 막고 있다 (그림 2 (B)). 하지만 현재까지 전사 신장 과정에서 히스톤 샤페론과 함께 뉴클레오솜 방어벽을 조정하는 크로마틴 리모델러가 존재할 것이라 예측해 왔지만 실제로 어떤 단백질이 이 역할을 수행하는지는 아직까지 밝혀지지 않고 있다. 또한 전사 종결 단계에서 RNA 중합 효소가 있게 되는 뉴클레오솜 자유 구간이 전사 종결에 어떠한 영향을 미치는지에 대해서도 연구가 이루어지지 않고 있다.
3. 결론
지금까지 본 글에서는 염색질의 뉴클레오솜 위치 지정이 어떻게 일어나는지, 그리고 뉴클레오솜 배열이 유전자의 발현에 어떻게 영향을 주는가를 살펴보았다. 염색질의 최소 단위이자 염색질의 구조와 특징을 결정하는데 핵심적인 후성 유전학적 요소인 뉴클레오솜은 히스톤 말단에서 일어나는 번역 후 변형들 및 이형 히스톤 단백질들에 의해 각 뉴클레오솜의 안정성 및 다른 단백질들 – 전사 요소들이나 주변의 뉴클레오솜들 – 과의 결합력이 달라진다. 그리고 DNA에 나열되어 있는 뉴클레오솜들은 DNA 염기 서열과 다양한 DNA 결합 단백질들, 그리고 그 단백질들로 인하여 생기는 뉴클레오솜 자유 구간 주변에서 위치 지정이 분명하게 되는 뉴클레오솜들과 크로마틴 리모델러 및 히스톤 샤페론들, 마지막으로 RNA 중합효소에 의해서 그 위치가 지정된다. 또한 이렇게 형성되는 뉴클레오솜 배열은 유전자 전사 과정에서 단순히 RNA 중합효소의 진행을 방해하는 뉴클레오솜 방어벽으로 작용할 뿐 아니라, 프로모터에서 전사 시작 복합체의 형성 및 전사 신장 복합체로의 전환을 돕기도 하고 유전자의 전사 지역에서 생성될 수 있는 비정상적인 전사 물질들로 인하여 세포 내 전사체의 균형을 망가지는 것을 막기도 한다. 그리고 이 RNA 중합체에 의한 유전자 전사에 관여하는 핵심 요소에는 히스톤 말단의 번역 후 변형들과 이형 히스톤 단백질들, 크로마틴 리모델러와 히스톤 샤페론들이 각자의 역할을 수행한다는 사실을 알 수 있었다. 하지만 지금까지 이루어진 염색질에서의 전사 연구들은 주로 전사 시작 기작과 뉴클레오솜 배열과의 관계를 다루어왔으며, 이루어지는 전사 신장과 종결과 뉴클레오솜 배열과의 관계에 대해서는 연구가 많이 필요한 실정이다. 특히 전사 신장 과정에서 필수적인 뉴클레오솜 방어벽을 낮추는 일을 히스톤 샤페론들과는 달리 독립적으로 수행하는 크로마틴 리모델러가 존재하는지, 그리고 전사 종결 위치 하류에 진화학적으로 대단히 잘 유지되어 있는 뉴클레오솜 자유 구간이 유전자 전사 과정에서 어떠한 역할을 수행하며 이를 조절하는 후성 유전학적인 요소들에는 무엇이 있는지 밝혀내는 일들은 앞으로 우리들이 그 답을 찾아가야 할 질문들이다.
참고 문헌
| 1. | Almer, A., Hörz, W. (1986) Nuclease hypersensitive Regions with adjacent positioned nucleosomes mark the gene boundaries of the PHO5/PHO3 locus in yeast. EMBO J. 5:2681-87. |
| 2. | Anderson, J.D., Widom, J. (2000) Sequence and position-dependence of the equilibrium accessibility of nucleosomal DNA target sites. J Mol Biol 296:979-87. |
| 3. | Anderson, J.D., Widom, J. (2001) Poly(dA-dT) Promoter elements increase the equilibrium accessibility of nucleosomal DNA target sites. Mol Cell Biol 21:3830-39. |
| 4. | Basehoar, A.D., Zanton, S.J., Pugh, B.F. 2004. Identification and distinct regulation of yeast TATA boxcontaining genes. Cell 116:699.709 |
| 5. | Boeger, H., Griesenbeck, J., Kornberg, R.D. (2008) Nucleosome retention and the stochastic nature of promoter chromatin remodeling for transcription. Cell 133:716-26. |
| 6. | Brogaard, K., Xi, L., Wang, J-P., Widom, J. (2012) A map of nucleosome positions in yeast at base-pair resolution. Nature 486:496-501. |
| 7. | Hodges, C., Bintu, L., Lubkowska, L., Kashlev, M., Bustamante, C. (2009) Nucleosomal fluctuations govern the transcription dynamics of RNA polymerase II. Science 325:626-28. |
| 8. | Kaplan, N., Moore, I.K., Fondufe-Mittendorf, Y., Gossett, A.J., Tillo, D., et al. (2009) The DNA-encoded nucleosome organization of a eukaryotic genome. Nature 458:362-66. |
| 9. | Korber, P., Hörz, W. (2004) In vitro assembly of the characteristic chromatin organization at the yeast PHO5 promoter by a replication-in dependent extract system. J Biol Chem. 279:35113-20. |
| 10. | Kornberg, R.D., Stryer, L. (1988) Statistical distributions of nucleosomes: nonrandom locations by a stochastic mechanism. Nucleic Acids Res. 16:6677-90. |
| 11. | Lomvardas, S., Thanos, D. (2002) Modifying gene expression programs by altering core promoter chromatin architecture. Cell 110:261–71. |
| 12. | Luger, K., Mader, A.W., Richmond, R.K., Sargent, D.F., Richmond, T.J. (1997) Crystal structure of the nucleosome core particle at 2.8 Å resolution. Nature 389:251–60. |
| 13. | Ramirez-Carrozzi, V.R., Braas, D., Bhatt, D.M., Cheng, C.S., Hong, C., et al. (2009) A unifying model for the selective regulation of inducible transcription by CpG islands and nucleosome remodeling. Cell 138:114–28. |
| 14. | Rhee, H.S., Pugh, B.F. (2012). Genome-wide structure and organization of eukaryotic pre-initiation complexes. Nature 483:295–301. |
| 15. | Valouev, A., Johnson, S.M., Boyd, S.D., Smith, C.L., Fire, A.Z., Sidow, A. (2011) Determinants of nucleosome organization in primary human cells. Nature 474:516-20. |
| 16. | Whitehouse, I., Rando, O.J., Delrow, J., Tsukiyama, T. (2007) Chromatin remodeling at promoters suppresses antisense transcription. Nature 450:1031-35. |
| 17. | Zhang, Z., Wippo, C.J., Wal, M., Ward, E., Korber, P., Pugh, B.F. (2011) A packing mechanism for nucleosome organization reconstituted across a eukaryotic genome. Science 332:977-80. |
첨부파일