
생화학분자생물학회입니다.
단백질 N-인산화 연구의 최근 동향
작성자
관리자작성일자
2018-12-01조회수
1316
.jpg)
1. 서론
단백질 인산화는 타깃 단백질의 기능을 조절하는 핵심적인 기작 중 하나로서, 기초과학뿐 아니라 의학 및 약학에서도 큰 의미가 있으며, 실제로 단백질 인산화와 탈인산화를 조절하는 인산화효소와 탈인산화효소는 중요한 신약개발의 타깃으로, 백혈병 치료제인 글리벡(Gleevec®)으로 대표되는 수십여 종의 인산화효소 저해제가 암과 당뇨병을 비롯한 여러 질병 치료에 사용되고 있다.
지금까지의 단백질 인산화 연구는 pSer, pThr, pTyr 등 3가지 형태의 단백질 O-인산화에 집중되어 있다 (그림 1). 이들 O-인산화 아미노산은 화학적으로 안정한 인산화 에스터로서 그 연구 방법론이 잘 확립되어 있으며, Ser, Thr, Tyr에 작용하는 인산화효소와 탈인산화효소에 관한 연구 또한 매우 활발하게 진행되고 있다. 그러나 생체 내에는 인산화 히스티딘(Phosphohistidine, pHis) (1), 인산화 라이신(Phospholysine, pLys), 인산화 아르기닌(Phosphoarginine, pArg) (2)과 같은 단백질의 N-인산화 현상도 존재한다. 이들 N-인산화는 이미 1950-60년대에 발견되었고 (pTyr는 1981년에 발견), 세포 신호 전달, 암세포 대사와 같은 현상에 관여한다는 것이 밝혀졌으나, pSer/pThr/pTyr 연구에 비하면 그 후속 연구는 상대적으로 부진한 상황이다. 이는 단백질의 N-인산화가 중요하지 않기 때문이라기보다는 연구 방법의 어려움 때문에 연구자들이 쉽게 접근하지 못하였기 때문이라고 볼 수 있다. 일례로 P. polycephalum 에서는 전체 인산화 단백질의 6%가 pHis인 것으로 밝혀졌는데, pTyr가 인산화 단백질의 1% 정도에 불과한 것을 감안하면 pHis와 같은 단백질 N-인산화는 우리의 생각보다 흔하게 생체 내에 존재함을 추정할 수 있다.
단백질 N-인산화와 기존에 연구된 O-인산화의 가장 큰 차이점은 그 화학적 안정성이다. 단백질 N-인산화의 산물인 pHis, pLys, pArg는 화학적으로 포스포아미데이트(Phosphoramidate)[1]의 형태로서, pSer, pThr, pTyr보다 훨씬 불안정하며 실제로 산성 조건하에서 쉽게 탈인산화되므로 그 연구가 기술적으로 매우 까다롭다. 이러한 불안정성 때문에 이들 N-인산화 현상을 선택적으로 검출할 수 있는 항체가 개발되지 못하였고 (항원이 생체 내에서 쉽게 분해), 산성 조건을 요구하는 기존의 LC-MS/MS 방법론도 바로 적용이 어려웠다. 그 결과, N-인산화 연구는 고전적인 방사성 동위원소(32P) 표지법에 의존하여 세포 내에서 어떤 단백질들이 pHis, pAsp, pLys 등을 포함하고 있는지도 파악이 쉽지 않았으며, N-인산화를 조절하는 인산화효소와 탈인산화효소에 관한 연구도 어려웠다. 따라서 최근 화학생물학적 방법론과 인산화 단백질 질량분석법의 비약적 발전이 있기 전까지 단백질 N-인산화 연구는 미개척 상태로 남아 있었다.
21세기 들어 질량분석법과 화학생물학적 방법론의 발전에 힘입어 이들 불안정한 N-인산화 현상에 대한 많은 새로운 발견이 이루어졌다. 그 결과 이들이 기존에 알려진 대사작용과 신호 전달에서뿐 아니라, Tumor Suppression과 암 전이, 그리고 Stress Response에서도 중요한 역할을 하는 것이 밝혀졌다. 또한 이들 현상을 매개하는 인산화효소와 탈인산화효소들도 새롭게 발견되어 기존의 단백질 인산화 연구의 새로운 방향성을 제시하고 있으며, 본 연구실에서도 His와 Arg의 인산화효소와 탈인산화효소 연구를 진행 중이다. 본 글에서는 최근의 발견을 중심으로 단백질 N-인산화 현상과 이를 조절하는 효소들의 생물학적 기능과 연구 방법론을 간략히 소개하고자 한다.
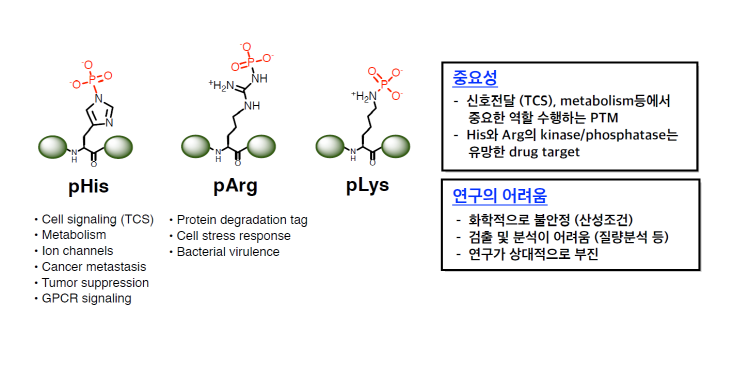
그림 1. 단백질 N-인산화의 예시
2. Phosphohistidine (pHis) (1)
pHis는 50여 년 전에 처음 발견되었으며, 세포 신호 전달 및 대사 과정에 핵심적으로 관여하는 것으로 알려져 있다. 이 중, 박테리아와 식물의 2요소 신호전달계(Two-Component Signaling System, TCS)은 센서 시스템으로서, 세포가 다양한 외부 환경의 변화를 감지하고 대응하는 데에 중요한 역할을 한다. TCS의 센서 단백질인 히스티딘 인산화효소(Histidine Kinase, HK)는 세포 외부 신호에 의해 자가인산화되어 pHis를 형성하고, 이 pHis가 전사인자인 Response Regulator(RR)를 활성화시켜 타깃 유전자 발현을 조절한다. 박테리아의 TCS는 병독성, 항생제 내성, 바이오 필름 등의 병리 현상을 조절하며, 식물의 TCS인 사이토키닌(Cytokinin) 신호전달 경로는 종자 발생 및 스트레스 저항성을 조절하므로 TCS 연구는 차세대 항생제의 개발과 농작물 저항성 강화 등에도 중요한 의미가 있다.
이뿐만 아니라 동물 세포에서는 암의 전이과정에 관여하는 NDPK, 해당작용(Glycolysis)와 암세포 대사에 관여하는 PGAM1와 ATP Citrate Lyase 등에서 pHis가 핵심적인 기능을 하며, 이들 단백질은 새로운 항암 요법의 타깃으로 연구가 진행 중이다. 또한 T 세포의 활성화에 관여하는 KCa3.1를 비롯하여 TRPV5와 같은 이온 채널들도 pHis에 의해 그 기능이 조절된다. 이 밖에 Heterotrimeric Gbeta 단백질과 히스톤 H4는 pHis가 GPCR 신호전달과 후성유전학(Epigenetics)에도 관여함을 시사한다.
위의 연구들은 방사성 동위원소 표지법에 의한 고전적 생화학 연구를 통해 어렵게 진행되었으나, 최근 pHis에 선택적인 항체 개발 (3)과 질량분석법의 발전에 힘입어, pHis 단백질과 그 인산화효소 및 탈인산화효소 연구에 많은 발전이 있었다. 특히 pHis의 프로테오믹스 연구 결과 대장균 (4, 5)과 암세포 (6)에서 기존의 예상을 뛰어넘는 수많은 종의 pHis 단백질이 존재함이 밝혀졌다. 아직 이들 단백질에서의 pHis의 역할은 정확히 규명되지 않았으나, 이처럼 많은 pHis 단백질이 존재한다는 사실만으로도 기존 단백질 인산화 연구와는 다른 새로운 방향의 미개척 분야의 가능성을 제시하고 있다고 하겠다.
이러한 히스티딘 인산화를 매개하는 히스티딘 인산화효소는 박테리아와 식물의 TCS에서 가장 잘 알려져 있으나, 동물 세포의 경우 NDPK 단백질이 유일한 예로 밝혀져 있다. NDPK는 원래 암세포의 전이 억제인자(Metastasis Suppressor)로 알려졌고, 세포 내의 NDP를 NTP로 인산화시키는 역할을 하나, KCa3.1, TRPV5 등의 이온채널과 ATP Citrate Lyase, Gbeta 단백질의 His 잔기를 인산화시킴으로서 그 기능을 활성화 또는 저해하는 것으로 알려져 있다. 그러나 지금까지 알려진 대다수 pHis 단백질의 경우에는 그 인산화효소가 알려져 있지 않으며, 이를 밝히는 것이 앞으로 pHis 연구의 중요한 과제가 될 것이다.
또한 pHis 잔기를 탈인산화시키는 pHis 탈인산화효소의 경우, 사람에서는 PHPT1, LHPP, 그리고 PGAM5가 알려져 있다. PHPT1은 KCa3.1, TRPV5와 같은 이온 채널과 ATP Citrate Lyase의 pHis를 탈인산화시키며, 그 결과 이들 단백질을 비활성화시킨다. 또한 LHPP는 우울증과 고환암 등의 유전적 위험인자로 알려져 왔으나, 2018년에 Hindupur 등이 LHPP가 간암세포에서 종양 억제인자로 작용함을 발표하여, 그 기능이 더욱 주목받게 되었다 (7). 특히 실제 환자 샘플에서 LHPP의 발현량이 매우 낮음을 확인하였고, 암 세포주에 LHPP를 발현시킬 경우 세포 사멸이 관찰되어 앞으로 새로운 항암 요법의 후보가 될 수 있음을 시사하였다. 마지막으로 PGAM5는 Histidine 인산화효소인 NDPK-B를 탈인산화하고, 그 결과로 KCa3.1의 탈인산화와 비활성화를 유도함으로써 CD4+ T 세포의 활성화를 막는 것으로 밝혀졌다. 이처럼 pHis 탈인산화효소들의 다양하고 중요한 생물학적 기능들이 속속 밝혀지고 있으나, 아직 밝혀지지 않은 탈인산화효소의 존재 여부 및 이들 효소의 pHis 기질 범위 등은 아직 미지수이다.
|
N-인산화 |
해당 단백질 및 그 생물학적 기능 | |
|
pHis |
pHis |
· Histone H4 (Epigenetics?) · Ion Channels (KCa3.1, TRPV5) · Heterotrimeric Gbeta Protein (GPCR Signaling) · > 200 proteins in E. coli, 768 Proteins in Human Cancer Cells |
|
Histidine |
· Two-Component System (박테리아와 식물 Cell signaling) · NME/NDPK (Tumor Suppressor) – Phosphorylates KCa3.1, TRPV5, Heterotrimeric Gbeta, and ATP Citrate Lyase | |
|
pHis |
· PHPT1 (Cancer Metastasis, Cell Skeleton조절) – Dephosphorylates KCa3.1, TRPV5, ATP Citrate Lyase · LHPP (Tumor Suppressor, Major Depression Disorder) · PGAM5 – Dephosphorylates NDPK-B. | |
|
pArg |
pArg |
· Hundreds of Proteins in B. subtilis 및 S. aureus (Protein Degradation Tag) · Histone H3? |
|
Arginine |
· McsB (Stress Response, Protein Degradation in Gram-(+) Bacteria) · Mammalian Arg Kinase Activities for Histones H3 (Uncharacterized) | |
|
pArg |
· YwlE (Stress Responses in B. subtilis) · PtpB (Stress Response in S. aureus) · CG31469 (D. melanogaster. Unknown Functions) | |
|
pLys |
pLys |
· Histone H1? |
|
Lysine |
· Unknown | |
|
pLys |
· PHPT1, LHPP | |
표 1. N-인산화를 포함한 단백질 및 그 인산화효소와 탈인산화효소
3. Phosphoarginine (pArg) (2)
pArg(Phosphoarginine, pArg)은 Arg의 구아니딘기가 인산화된 형태로, pArg 아미노산 자체는 척추동물의 Creatine처럼 무척추동물의 근육 내에서 화학적 에너지를 저장하는 포스파겐(Phosphagen)의 역할을 한다. 1970년대에 단백질 내의 Arg 잔기 또한 인산화되어 pArg를 형성하는 것이 발견되었으나, pArg의 화학적 불안정성 때문에 후속 연구에 많은 어려움이 있었다. 그러나 21세기에 들어서 pArg 연구에 새로운 화학생물학적 도구가 개발되고 질량분석법이 발달하면서, 그 생물학적인 기능이 점차 밝혀지기 시작하였다. 특히 박테리아에서는 단백질의 pArg 잔기가 단백질 분해 신호 등의 중요한 역할을 한다는 것이 밝혀졌다.
생체 내에서 어떤 단백질이 pArg를 포함하고 있는지 알아내기 위해서는, 세포로부터 얻은 단백질 시료로부터 pArg을 포함한 단백질 또는 펩타이드를 선택적으로 분리하는 것이 필수적이다. 그러나, 일반적으로 인산화 펩타이드의 선택적 분리에 쓰이는 IMAC 방법은 산성 조건에서 진행되므로 pArg에 적용이 어려웠다. 또 다른 방법은 pArg에 선택적으로 결합하는 항체를 이용한 면역침강법(Immunoprecipitation)인데, pArg 자체를 항원으로 사용하면 토끼 등의 동물 체내에서 쉽게 분해되면서 항체가 얻어지지 않는 문제가 있었다. 이를 극복하기 위해 2015년에 Thompson 연구실에서 pArg 항원의 분해를 막을 수 있도록 pArg의 불안정한 인-질소 결합을 상대적으로 안정한 인-탄소 결합으로 치환한 pArg 모방체를 설계 및 합성하였다. 그리고 이 모방체를 항원으로 이용함으로써 마침내 pArg 선택적 항체를 개발하는데 성공하였다.
이처럼 화학자들이 pArg 연구의 새로운 방법론을 개발하는 것에 맞물려 생물학자들 또한 pArg의 새로운 기능에 관한 실마리를 발견하기 시작하였다. 그람 양성균 Bacillus subtilis의 McsB 단백질은 본래 Tyrosine 인산화효소로 알려져 있었다. 또한 McsB는 포스파겐 인산화효소와 유사한 활성부위를 포함하고 있다는 사실 또한 이전부터 알려져 있었다. 2009년에 Clausen 그룹은 질량분석 및 단백질 구조분석을 통하여 McsB가 사실은 Arg 인산화효소로 작용한다는 것을 발견하여 Science지에 보고하였다 (8). 이는 생체 내에서 Arg 인산화효소의 실체와 기능이 정확하게 확인된 최초의 예로서 pArg 연구의 기념비적 논문이라 하겠다.
이 연구에 힘입어, 여러 그룹에서 pArg에 관한 후속 연구가 진행되었다. 특히 pArg 단백질의 질량분석과 프로테오믹스 연구 방법론이 개발되면서 많은 발견이 이루어졌다. 2012년에는 Gerth 그룹이 (9), 2014년에는 Clausen 그룹이 각각 Bacillus subtilis에서 수백여 개의 Arg 인산화 위치를 밝혀냈다 (10). 해당 연구를 통하여, Arg 인산화와 pArg 탈인산화효소 YwlE는 박테리아의 스트레스 대응 체계에서 중요한 역할을 한다는 것이 밝혀졌다. 또한 병원균인 S. aureus에서도 pArg 단백질이 다수 발견되었으며, YwlE의 Homolog인 PtpB가 비슷한 역할을 함이 밝혀졌다 (11).
2016년에는 Clausen 그룹에서 pArg이 진핵 세포에서의 단백질 분해 표식인 유비퀴틴(Ubiquitin)과 유사한 역할을 한다는 것을 발견하였다 (12). 즉, McsB 인산화효소에 의하여 생성된 pArg이 박테리아에서의 단백질가수분해효소 복합체인 ClpC-ClpP에 의해 인식됨으로써 pArg을 포함한 단백질의 분해가 일어나게 된다. 이 연구는 기존의 원핵세포의 단백질 분해 메커니즘과는 다른 pArg 표지(Tagging)를 이용한 새로운 메커니즘을 밝혀냈다는 점에서 생물학적으로 큰 의미를 가진다고 볼 수 있다. 또한 ClpP 단백질 가수분해효소는 S. aureus와 M. tuberculosis의 병독성을 조절하는 것으로 알려져 있어, 차세대 항생제의 타깃으로 활발히 연구되고 있다는 점에서 pArg의 연구는 의 약학적으로도 의미를 지닌다고 하겠다.
진핵 세포에서의 pArg의 기능은 훨씬 더 많은 부분이 미지의 영역에 남아있으며 어떠한 단백질이 pArg를 포함하고 있는지조차 파악되어 있지 않다. 다만 초파리에서는 YwlE의 상동유전자(Homolog)인 CG31469 단백질이 pArg 탈인산화효소로 기능함이 밝혀졌으나, 그 생물학적 역할은 미지수이다 (13). 또한 생쥐의 백혈병 세포 핵으로부터 히스톤 H3의 Arg를 인산화시키는 Arg 인산화효소가 발견되었으나, 후속 연구는 진행되지 않았다 (14). 다만 위에서 서술한 박테리아에서의 pArg 연구 방법론이 진핵 세포에 확장되어 적용될 경우, 새로운 발견으로 이어질 가능성이 있다.
4. Phospholysine (pLys) (2)
다른 단백질 N-인산화(pHis, pArg)에 비해서도 pLys는 훨씬 연구가 덜 되어 있다. 1970년 대에 재생 중인 쥐의 간 및 Walker256 암세포에서 히스톤 H1이 pLys를 포함하고 있는 것이 발견되었으나, 이 현상을 매개하는 인산화효소 및 그 생물학적 역할은 아직 자세히 밝혀지지 않았다. 다만 pHis 탈인산화효소로 알려진 PHPT1와 LHPP가 pLys 또한 가수분해하는 것으로 알려져 있으므로 이들 단백질의 생물학적 기능이 pLys와 연관되어 있는지 주목할 만하다.
5. 결론
역사적으로 볼 때 pHis를 비롯한 단백질 N-인산화 현상은 pTyr보다도 먼저 발견되었으나, 그 화학적 불안정성과 후속 연구의 어려움으로 인하여 그 생물학적 기능 및 인산화효소, 탈인산화효소 등이 상당수 밝혀지지 않은 채 남아 있다. 필자의 경우, pHis에 선택적인 항체의 개발로부터 시작하여 이들 불안정한 단백질 인산화 현상을 계속 연구해 오고 있는데 (1, 3, 15), 최근 몇 년간 이들 ‘비전형적’인산화 현상에 대한 연구 결과가 점점 많이 발표되는 현상이 매우 고무적이라고 생각한다. 그리고 아직 상대적으로 미개척 상태로 남아있는 이 분야가 앞으로 더욱 많은 연구가 진행됨으로써 단백질 인산화 연구에 새로운 방향을 제시할 수 있게 되기를 기대하면서 부족한 소개글을 마칠까 한다.
6. 참고문헌
1. Kee, J.-M., Muir, T. W. (2012) Chasing Phosphohistidine, an Elusive Sibling in the Phosphoamino Acid Family. ACS Chem Biol 7 (1), 44–51.
2. Besant, P., Attwood, P., Piggott, M. (2009) Focus on Phosphoarginine and Phospholysine. Curr Protein Pept Sci 10 (6), 536–550.
3. Kee, J.-M., Oslund, R. C., Perlman, D. H., Muir, T. W. (2013) A Pan-Specific Antibody for Direct Detection of Protein Histidine Phosphorylation. Nat Chem Biol 9 (7), 416–421.
4. Potel, C. M., Lin, M.-H., Heck, A. J. R., Lemeer, S. (2018) Widespread Bacterial Protein Histidine Phosphorylation Revealed by Mass Spectrometry-Based Proteomics. Nat Methods 15 (3), 187–190.
5. Oslund, R. C., Kee, J.-M., Couvillon, A. D., Bhatia, V. N., Perlman, D. H., Muir, T. W. (2014) A Phosphohistidine Proteomics Strategy Based on Elucidation of a Unique Gas-Phase Phosphopeptide Fragmentation Mechanism. J Am Chem Soc 136 (37), 12899–12911.
6. Fuhs, S. R. et al. (2015) Monoclonal 1- and 3-Phosphohistidine Antibodies: New Tools to Study Histidine Phosphorylation. Cell 162 (1), 198–210.
7. Hindupur, S. K. et al. (2018) The Protein Histidine Phosphatase LHPP Is a Tumour Suppressor. Nature 555 (7698), 678–682.
8. Fuhrmann, J. et al. (2009) McsB Is a Protein Arginine Kinase That Phosphorylates and Inhibits the Heat-Shock Regulator CtsR. Science 324 (5932), 1323–1327.
9. Elsholz, A. K. W. et al. (2012) Global Impact of Protein Arginine Phosphorylation on the Physiology of Bacillus Subtilis. Proc Natl Acad Sci USA 109 (19), 7451–7456.
10. Schmidt, A. et al. (2014) Quantitative Phosphoproteomics Reveals the Role of Protein Arginine Phosphorylation in the Bacterial Stress Response. Mol Cell Proteomics 13 (2), 537–550.
11. Junker, S. et al. (2018) Spectral Library Based Analysis of Arginine Phosphorylations in Staphylococcus Aureus. Mol Cell Proteomics 17 (2), 335–348.
12. Trentini, D. B . et al. (2016) Arginine Phosphorylation Marks Proteins for Degradation by a Clp Protease. Nature 539 (7627), 48–53.
13. Fuhrmann, J. et al. (2013) Structural Basis for Recognizing Phosphoarginine and Evolving Residue-Specific Protein Phosphatases in Gram-Positive Bacteria. Cell Rep 3 (6), 1832–1839.
14. Wakim, B. T., Aswad, G. D. (1994) Ca(2+)-Calmodulin-Dependent Phosphorylation of Arginine in Histone 3 by a Nuclear Kinase From Mouse Leukemia Cells. J Biol Chem 269 (4), 2722–2727.
15. Jung. H., Shin, S. H., Kee, J.-M. (2018) Recent updates on protein N-phosphoramidate hydrolases. ChemBioChem, in press. https://doi.org/10.1002/cbic.201800566
[1] 단백질 N-인산화와 기존에 연구된 O-인산화의 가장 큰 차이점은 그 화학적 안정성이다. 단백질 N-인산화의 산물인 pHis, pLys, pArg는 화학적으로 포스포아미데이트(Phosphoramidate)의 형태로서, pSer, pThr, pTyr보다 훨씬 불안정하며, 실제로 산성 조건하에서 쉽게 탈인산화되므로 그 연구가 기술적으로 매우 까다롭다. 이러한 불안정성 때문에, 이들 N-인산화 현상을 선택적으로 검출할 수 있는 항체가 개발되지 못하였고 (항원이 생체 내에서 쉽게 분해), 산성 조건을 요구하는 기존의 LC-MS/MS 방법론도 바로 적용이 어려웠다.
첨부파일