
생화학분자생물학회입니다.
종양 연구에서 단일 세포 분석 (Single Cell Analysis in Cancer Biology)
작성자
관리자작성일자
2017-08-01조회수
581종양 연구에서 단일 세포 분석
(Single Cell Analysis in Cancer Biology)

주경민
성균관대학교 의과대학 해부세포생물학교실
kmjoo@skku.edu
1. 서론
암을 정복하고자 하는 현대 의학의 노력은 수십 년 동안 지속되어 왔다. 특히 항암화학치료(Chemotherapy) 분야는 분열하는 세포에 비특이적으로 작용하는 고전적 항암제로부터 시작하여 암세포에 존재하는 치료 표적(Specific Therapeutic Target)에 특이적으로 작용하는 표적항암제(Targeted Therapy), 항암면역반응을 유발하는 면역치료제(Immunotherapy)에 이르기까지 눈부신 발전을 거듭해왔다. 그러나 항암화학치료가 암환자의 생존기간을 늘리고 완치율을 높이는데 크게 기여했음에도 불구하고 여전히 암은 현대 의학이 정복해야 하는 영역으로 남아 있다. 새로운 항암화학치료제가 개발되고 적용될 때마다 환자 별로 효과가 있는 환자와 효과가 없는 환자가 존재하고, 유의한 효과가 있더라도 많은 비율의 환자에서 내성(Resistance)이 발생하는 점을 고려해보면 앞으로 항암화학치료는 항암화학치료제 별로 효과를 보일 것으로 예상되는 환자들을 선별하여 치료하는 개인 맞춤치료(Personalized Treatment)와 항암화학치료 내성을 예방하거나 극복할 수 있는 병용항암화학치료(Combinational Treatment)가 활발히 연구될 것으로 예상된다.
이 중 병용항암화학치료는 결핵(Tuberculosis) 치료와 유사성이 있다. 현재 결핵균을 죽일 수 있는 항생제가 다수 개발되어 있지만 결핵균은 큰 포식 세포(Macrophage) 안에서 생존하면서 돌연변이로 약제 내성을 획득하는 비율이 높다. 이 때문에 효능이 있는 항생제 여러 개를 동시에 투여함으로써(Isoniazid, Rifampicin, Ethambutol, Pyrazinamide 4가지 약물 동시 투여가 현재 표준 요법 임), 투여하는 약제에 동시에 내성을 획득하는 확률을 낮추는 전략을 취하고 있다. 단순히 50%의 확률로 한 가지 항생제에 대해 약재 내성을 획득한다고 가정할 때, 4가지 항생제에 동시에 내성을 획득할 확률은 (0.5)4, 6.25%로 낮아진다. 결핵 환자가 다수의 결핵균을 보유한 것과 같이, 한 환자의 암 속에는 매우 많은 수의 암세포가 존재하고 이 암세포들은 각자 다른 항암화학치료제에 대해 높은 감수성을 가진다(종양 내 다양성, Intra-Tumoral Heterogeneity)고 가정하면 최대한 많은 암세포를 사멸시킬 수 있도록 항암화학치료제를 적절히 조합할 수 있다. 이때 실질적으로 어려운 점은 ‘암 속에 어떤 종류의 암세포가 존재하는지를 어떠한 방법으로 분석하여 수많은 항암화학치료제를 적절히 맞추어 조합할 것인가’가 될 것이다.
최근까지 암은 세포(Cell) 단위가 아니라 조직(Tissue) 단위로 분석해왔다. 따라서 많은 암세포가 존재하는 암조직을 한꺼번에 유전적으로 분석하면 수백 개~수천 개 세포의 유전적 특성을 평균적으로 분석하게 된다. 예를 들면 A라는 유전자의 발현이 높은 암세포 50개와 유전자의 발현이 낮은 암세포 50개가 섞여 있는 암조직을 분석하면 A 유전자의 발현은 중간 정도로 나타난다. 항암화학치료 감수성도 마찬가지일 것이다. B라는 항암화학치료제에 감수성이 높은 암세포 50개와 낮은 암세포 50개가 섞여 있는 경우, B 항암화학치료제의 감수성은 중간 정도로 분석되어 B 항암화학치료제는 암세포의 반을 사멸시킬 수 있음에도 불구하고 치료제로 선택되지 않을 것이다. 결국 다양한 감수성을 가진 암세포가 섞여 있는 암에서 최대한 많은 암세포를 사멸시킬 수 있도록 최적의 항암화학치료제를 조합하기 위해서는 암을 조직 단위가 아니라 세포 단위로 분석함으로써 암세포의 구성 현황을 알아내야 한다. 그동안 단일 세포(Single Cell) 단위의 분석은 기술적 어려움으로 인해 실현되기 어려웠으나 최근 기술적 난제가 서서히 해결되고 있다.
2. 단일 세포 유전자 발현 분석 기술
단일 세포 유전자 발현 분석 기술은 다시 말해 단일 세포 내 mRNA의 구성을 분석하는 것이다. 단일 세포 내 mRNA의 구성을 분석하기 위하여 1) 단일 세포를 분리, 2) 단일 세포 내의 mRNA를 분리, 3) 분리한 mRNA를 증폭, 4) 증폭한 mRNA의 염기서열 분석(mRNA Sequencing), 5) 염기서열의 의료정보학적 분석(Bioinformatic Analysis) 단계를 거치게 된다. 이 중 4) mRNA 염기서열 분석과 5) 염기서열의 의료정보학적 분석은 기존의 Next Generation Sequencing(NGS) 기술을 이용한 mRNA 염기서열 분석과 대동소이하다. 결국 단일 세포 유전자 발현 분석 기술에서 1) mRNA를 손상시키지 않은 상태에서 분석해야 하는 세포들을 단일 세포 단위로 분리하는 단계와 2~3) 단일 세포 내에서 mRNA를 분리하고 증폭하는 단계가 핵심이라고 할 수 있다. 여러 단일 세포로부터 연구자가 직접 손으로 안정적으로 일정하게 mRNA를 분리하고 증폭하는 것은 현실적으로 불가능하기 때문에, 이 단계를 자동으로 진행해주는 연구 장비와 시약 세트가 이미 개발되어 판매되고 있다. 대표적인 것이 Fluidigm 사의 C1 System과 10X Genomics 사의 Chromium System이다.
C1 system의 경우 한 개의 세포가 들어가는 크기의 홈에 음압(Negative Pressure)으로 세포를 잡아 단일 세포를 분리한 뒤, 세포막을 파괴하고 mRNA를 분리하는 Reaction Chamber로 세포를 밀어 넣는 방법을 취한다 (1). 10X Genomics 기기는 유세포분석기(Florescence Activated Cell Sorting, FACS)와 같이 시약 한 방울에 세포 한 개가 들어가도록 한 뒤, 이곳에서 세포막 파괴, mRNA 분리, mRNA 증폭이 연속적으로 이루어지도록 한다 (2). 두 시스템 모두 mRNA 증폭은 Oligo-dT Primer를 이용한 역전사(Reverse Transcription)로 진행되기 때문에 역전사 과정에서 mutation이 발생하기도 하고, mRNA의 종류와 양에 따라 증폭에 불균형이 일어나기도 한다. 따라서 mRNA 증폭과정에서 단일 세포의 mRNA 구성이 잘 유지되는지를 반드시 검증해주어야 한다. 또한 역전사 효소(Reverse Transcriptase)의 오류로 인한 무작위 돌연변이(Random Mutation) 때문에 단일 세포 유전자 발현 분석을 통해서 단일 세포가 가지고 있는 돌연변이(Genetic Mutation)를 분석하는 것은 한계가 있다. 단일 세포에서 분리하는 mRNA의 숫자는 한정적이기 때문에 한 세포에서 분리, 증폭한 mRNA를 따로 염기서열 분석(NGS)하지 않고, 여러 세포의 mRNA를 섞어서 한꺼번에 분석하게 되는데 어느 세포에서 유래한 mRNA인지를 구분하기 위하여 Oligo-dT Primer에 세포를 구분할 수 있는 짧은 염기서열(Oligonucleotide, Bar Code)을 붙여 Primer를 제작함으로써 문제를 해결하고 있다.
이처럼 몇 가지 한계점에도 불구하고 단일 세포에서 mRNA를 분리, 증폭, 염기서열 분석하는 기술은 비교적 잘 확립되어 있다. 자동화되지 못한 채 전통적인 연구자의 기술에 의존하고 있으며 단일 세포 유전자 발현 분석 결과에 많은 영향을 미치는 부분은 결국 단일 세포 분리 기술이다. 조직에서 세포를 분리하면 세포 이외에 다량의 세포 바깥 물질(Extracellular Matrix)이 섞이게 되고 세포 바깥 물질이 세포 크기와 유사한 형태로 섞여 있는 상태에서는 이후에 진행되는 단일 세포 유전자 발현 분석 모든 과정의 효율이 크게 떨어질 수밖에 없다. 또한 세포를 살아있는 상태로 분리하는 것이 중요한데 세포가 사멸하거나 사멸한 상태가 되면 세포 내 mRNA의 상태와 구성이 크게 변화하기 때문이다. mRNA 분리, 증폭, 염기서열 분석과 같이 표준화된 기법이 정립된다면 도움이 되겠으나 조직의 종류와 상태에 따라 단일 세포 분리 방법을 최적화해야 한다는 한계점이 있다. 이와 같은 이유로 조직의 세포를 직접 분리하는 것보다 배양된 세포로부터 연구를 시작하는 것이 세포 분리 과정에서의 어려움을 줄일 수 있다.
3. 암조직의 단일 세포 유전자 발현 분석
암조직으로부터 물리적/화학적 방법을 이용해 단일 세포들을 분리한 후, 단일 세포 유전자 발현 분석 기술을 이용하여 세포들의 유전자 발현을 각각 분석해보면 유전자 발현 특성에 따라 암세포와 암조직 내 정상세포(혈관내피세포, 섬유모세포, 면역세포 등)를 구분할 수 있으며, 암세포만을 따로 분리하여 암세포들의 유전자 발현 특성(다양성)을 분석할 수 있다. 실제로 하나의 암조직으로부터 분리한 암세포들이라고 하더라도 그 유전자 발현 특성은 무척 다양하다. 중요한 점은 오랜 시간 동안 in vitro 배양을 거쳐 확립된 암세포주(Cell Line)보다 암환자의 암조직으로부터 바로 분리한 암세포들의 유전자 발현 다양성이 월등히 높다는 점이다. 따라서 암세포의 다양성을 분석하기 위해서는 암세포주를 이용하는 것은 지양하는 것이 바람직하다. 반대로 환자의 암조직으로부터 직접 단일 세포를 분리하면, 암조직의 특성상 괴사(Necrosis)된 부분이 많이 섞여 있고 암세포 외에 다수의 정상세포(혈관내피세포, 섬유모세포, 면역세포 등)가 존재한다. 괴사된 부분이 많이 섞여 있으면 암세포의 생존율이 떨어지고 암세포 외에 다수의 정상세포가 섞여 있으면 암세포를 높은 비율로 회수할 수 없다는 단점이 있다.
건강한(mRNA가 잘 보존된) 암세포를 최대한 높은 비율로 얻는 방법으로 이종이식 동물모델(Patient-Derived Xenograft(PDX),‘AVATAR’ Mouse)을 이용하는 방법이 있다. 암환자의 암조직으로부터 분리한 암세포를 면역력이 억제된(Immune-Deficient) 생쥐(Mouse)에 이식하면 상대적으로 단기간에(2~3개월) 많은 수의 암세포를 얻을 수 있다. ‘AVATAR’ Mouse를 이용하여 암세포를 증폭하더라도 원래 암조직의 유전적, 기능성 특성이 변하지 않고 유지된다는 점은 다수의 연구를 통해 입증되어 왔다 (3). 본 연구자의 선행연구 결과를 검토해보면 암세포의 유전자 발현 다양성도 이종이식 동물모델에서 잘 유지되었다 (4, 5). 이종이식 동물모델을 이용하면 암조직 내의 정상세포가 생쥐의 세포이므로 사람 암세포만을 비교적 쉽게 분리해 낼 수 있다. 다만 암조직 내의 정상세포의 유전자 발현도 동시에 분석하는 경우 이종이식 동물모델의 암미세환경(Microenvironment)은 생쥐 세포라는 점에서 그리고 면역세포가 결핍되어 있다는 점에서 약점이 있다. 반대로 동물모델을 이용하면 항암치료 전후의 암세포 유전자 발현 다양성 분석의 변화, 전이암과 원발암의 유전자 발현 다양성 차이 분석 등 보다 통제된 환경에서의 연구가 가능하다.
4. 암세포 유전자 발현 다양성을 이용한 병합항암화학치료 구성
하나의 암조직에서 암세포를 분리한 뒤, 각 암세포의 유전자 발현 특성을 단일 세포 유전자 발현 분석 기술을 통해 분석하면 각 세포의 유전자 발현 특성에 따라 그 세포가 감수성을 보일 것으로 예상되는 항암치료제를 예측할 수 있다. Epidermal Growth Factor Receptor(EGFR)가 활성화된 세포에서 관찰된 유전자 발현 특성과 같은 양상의 유전자 발현 특성을 가진 암세포는 EGFR 관련 신호전달체계(Signaling Pathway)가 활성화되어 있을 것으로 예상하고, 그 암세포는 EGFR 억제제(Inhibitor)에 감수성을 보일 것으로 예측하는 식이다. 일례로 한 신세포암(Renal Cell Carcinoma)에서 분리한 암세포들의 유전자 발현 특성을 단일 세포 단위로 분석하였을 때, EGFR 신호전달체계와 Src 신호전달체계가 활성화된 것으로 예상되는 암세포가 많다는 것을 관찰할 수 있었다. 이때 단일 세포 단위의 분석을 통하면 EGFR 신호전달체계와 Src 신호전달체계가 같은 세포에서 활성화된 것인지 아니면 상이한 세포에서 활성화된 것인지까지 분석할 수 있다. 만약 같은 세포에서 두 신호전달체계가 모두 활성화된 것이라면 두 신호전달체계를 동시에 억제해도 더 많은 수의 암세포 사멸을 유도하기 어려울 것이나 다른 세포에서 각각의 신호전달체계가 활성화된 것일 때에는 병합항암화학치료에 의해 더 많은 수의 암세포 사멸을 유도할 수 있을 것이다.
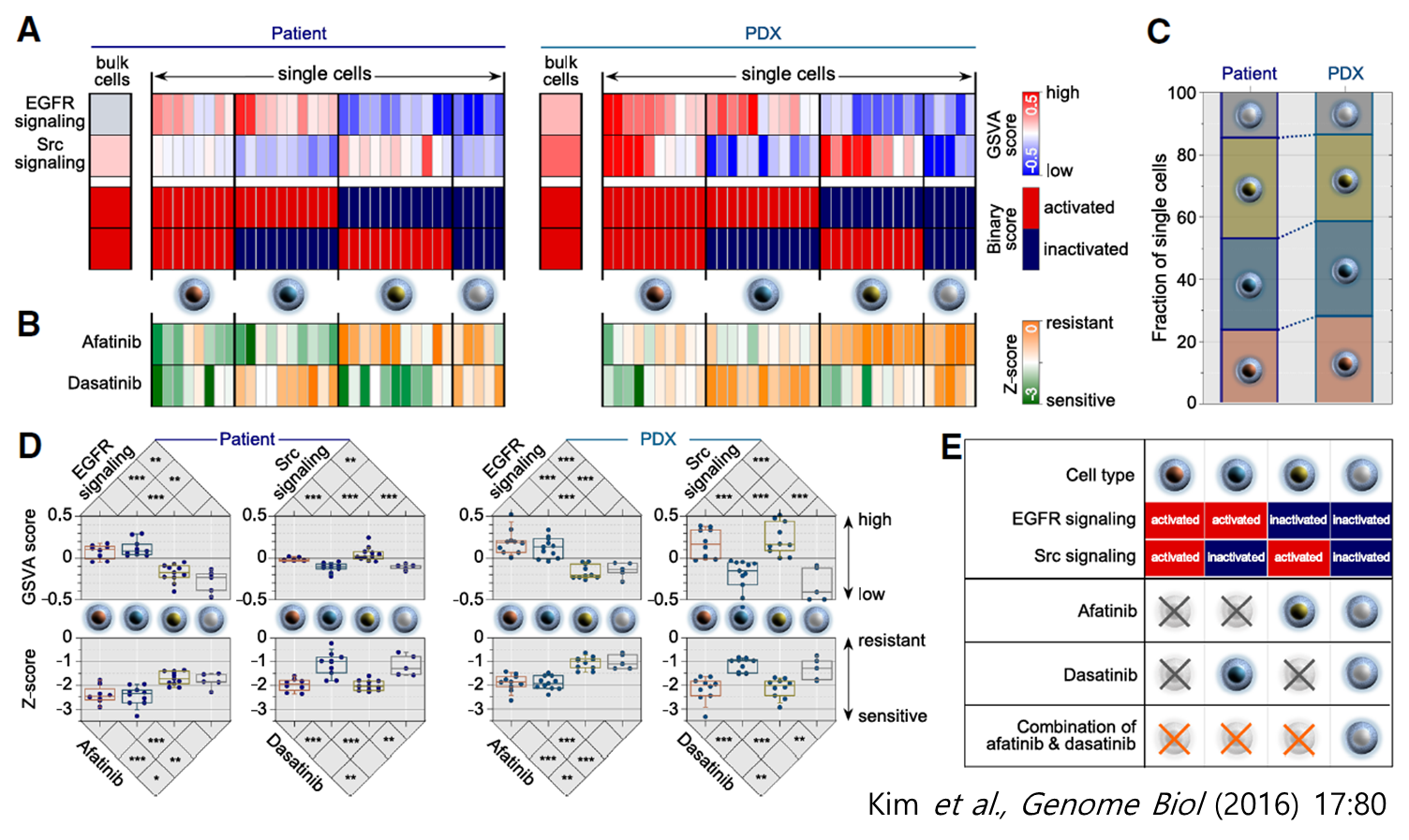
그림 1. 신세포암세포의 단일 세포 유전자 발현 분석을 통한 항암화학치료제 반응 예측 (4)
분석 결과, 이 신세포암에서 EGFR 신호전달체계와 Src 신호전달체계가 활성화된 것으로 예상되는 암세포는 일치하지 않는 것으로 나타났다 (그림 1). 이는 병합항암화학치료에 의해 더 많은 수의 암세포 사멸을 유도할 수 있을 것이라는 점을 시사하고 실제로 EGFR 억제제와 Src 억제제를 병합하여 사용하였을 때, 각각의 약제를 단독으로 사용하였을 때보다 치료 효과가 in vitro와 in vivo 모두에서 유의하게 높았다 (4). 신세포암세포의 유전자 발현 양상을 단일 세포 단위로 분석함으로써 감수성이 있는 항암화학치료제를 예측하고 병합 전략을 세울 수 있는 것이다. 이 연구에서는 실험적으로 예측한 항암화학치료제의 병합 치료 효과를 실제 임상적으로 확인하지는 못한 한계점이 있고 병합 치료를 시행하였을 경우 실제로 항암제 내성 발생 비율이 낮아지는지를 연구하지 못한 한계점이 있다. 앞으로 단일 세포 단위의 유전자 발현 분석에 더하여 약물반응과 관련된 유전적 변이들(예를 들어 Crizotinib에 대한 반응이 예상되는 비소세포성 폐암(Non-Small Cell Lung Cancer)에서의 EML4-ALK Fusion)을 함께 분석하여 반영한다면 단일 세포 유전자 발현 분석의 한계점을 보완하여 더 높은 정확성을 가진 병용항암화학치료 설계가 가능할 것으로 예측된다.
5. 종양 연구에서 단일 세포 분석의 발전 방향
여기서는 암세포의 단일 세포 유전자 발현 분석을 통한 병용항암화학요법 반응 예측에 대해 정리하였지만 단일 세포 분석의 응용 분야는 훨씬 넓을 것으로 예상된다. 단일 세포 단위의 유전자 발현 분석뿐만 아니라 유전적 변이나 후생적 변화(Epigenetic Change) 측면에서의 암세포 다양성을 분석할 수 있을 것이고 더 나아가서는 단일 세포 내에서 유전적 변이, 후생적 변화, 유전자 발현을 동시에 분석할 수 있는 기술도 개발 중이다. 암세포뿐만 아니라 암조직 내 정상 세포도 중요한 연구 대상이 되는데, 앞서 기술한 대로 암조직에서 세포를 분리하여 단일 세포 단위로 유전자 발현을 관찰하면 세포의 종류를 알아낼 수 있다. 즉 암조직 내 각 세포 종류마다 어떠한 유전자 발현 특성을 보이는지, 암 조직 내에서 그 세포 종류의 유전자 발현이 정상과 달리 어떻게 변화하는지를 분석할 수 있는 것이다. 암세포뿐만 아니라 암미세환경을 구성하는 정상 세포들이 암의 진행, 내성 발현, 특히 면역치료 반응을 조절/결정한다는 측면에서 이 정상 세포들은 과학적, 의학적으로 매우 높은 중요성을 가진다.
현재는 암조직에서 세포를 물리적/화학적 방법으로 분리할 때, 그 세포가 암조직의 어느 위치에서 유래하였는지에 대한 공간적 정보는 존재하지 않는다. 그러나 암조직 내 위치 정보와 유전자 발현을 단일 세포에서 함께 분석할 수 있다면, 주위 세포 사이의 상호작용(Interaction)까지 분석할 수 있을 것이다. 이처럼 단일 세포 분석 기술은 앞으로 계속 발전할 것이고, 여러 측면에서 종양 연구에 기여할 것으로 생각된다. 그동안 기술적 한계로 인해 제대로 밝혀지지 못했던 암세포의 다양성, 암조직 내 정상 세포의 특성 등에 대한 과학적 연구결과가 계속 축적된다면 현재 우리나라 사망원인 1위인 암의 정복에 한 발짝 더 다가설 수 있을 것으로 기대한다.
참고문헌
1. https://www.fluidigm.com/products/c1-system
2. https://www.10xgenomics.com/single-cell
3. Joo, K. M. et al. (2013) Patient-specific orthotopic glioblastoma xenograft models recapitulate the histopathology and biology of human glioblastomas in situ. Cell Rep 3, 260-273
4. Kim, K. T. et al. (2016) Application of single-cell RNA sequencing in optimizing a combinatorial therapeutic strategy in metastatic renal cell carcinoma. Genome Biol 17, 80.
5. Kim, K. T. et al. (2015) Single-cell mRNA sequencing identifies subclonal heterogeneity in anti-cancer drug responses of lung adenocarcinoma cells. Genome Biol 16, 127.
첨부파일