
생화학분자생물학회입니다.
Structural Biology of Macromolecular Interactions Laboratory (구조생물학 연구실)
작성자
관리자작성일자
2017-08-01조회수
572Structural Biology of Macromolecular Interactions Laboratory
(구조생물학 연구실)
이상호
성균관대학교 생명과학과
[연구실소개]
우리가 볼 수 있는 생명현상은 다양한 단백질 사이에서 일어나는 상호작용으로 이루어진 네트워크를 통해 나타난다. 즉, 생명현상을 이해하기 위해서는 단백질 간의 상호작용을 이해하는 것이 필수적이다. 그러나 단백질 네트워크를 연구하기 위해 이용되는 여러 생화학적 연구기법들은 표지를 통한 간접적인 방식이라는 한계가 있다. 따라서 단백질 간의 상호작용을 정확하게 이해하기 위해서는 직접 단백질의 구조를 살펴보고, 어떤 방식으로 상호작용이 일어나는지 연구할 필요가 있다.
본 연구실에서는 X-선 결정법(X-ray crystallography), 소각 X-선 산란(small-angle X-ray scattering), 극저온 전자현미경(cryo-electron microscopy) 등 다양한 기법을 통해 여러 단백질의 구조를 규명하고, 이를 통해 단백질 간의 상호작용을 이해하는 연구들을 다양한 생화학적, 생물리학적 기법을 이용하여 수행하고 있다. 그리고 대상 단백질이 실제 생명체의 단백질 네트워크 상에서 어떤 기능을 하는지 여러 세포생물학적 연구 기법들을 이용하여 연구하고 있다. 또한 단백질의 구조적 연구를 기반으로 질병의 진단 및 치료, 농업 등 응용분야에 적용하는 연구를 진행 중이다(그림 1).
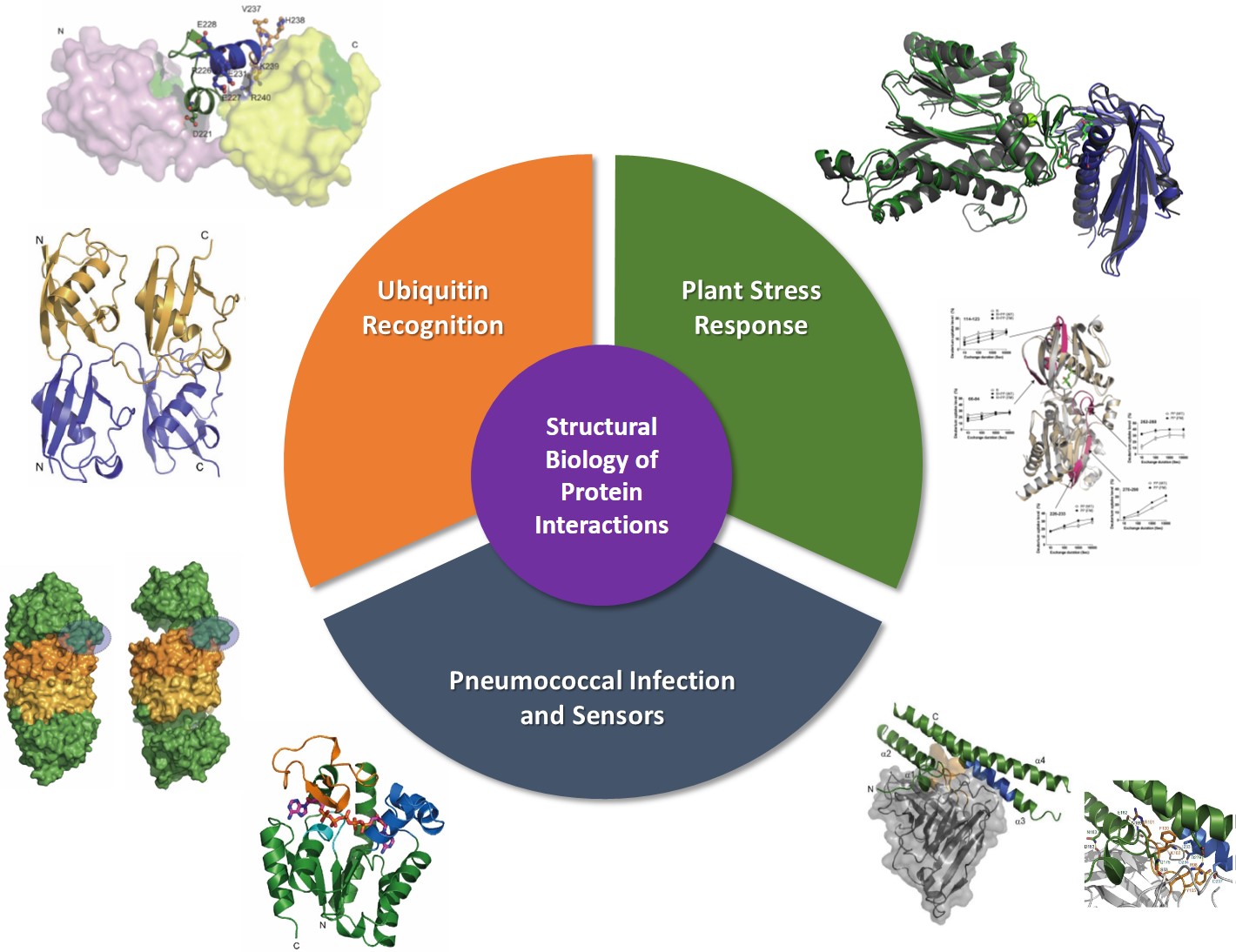
그림 1. 본 연구실에서 수행하고 있는 연구주제
[연구내용]
1. 유비퀴틴화에 의한 단백질 기능 조절
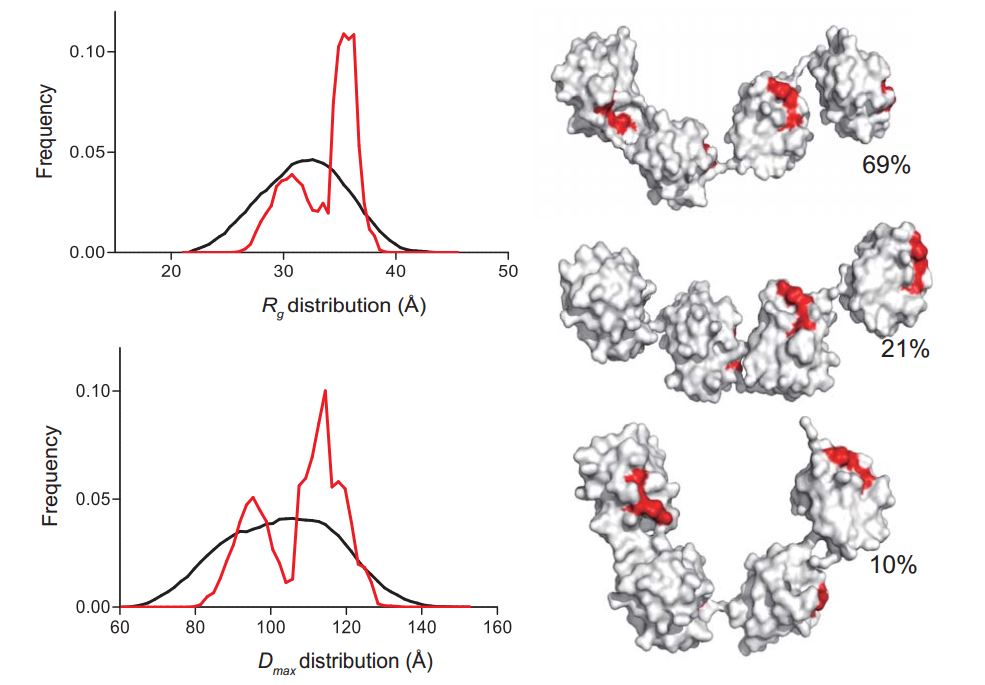
그림 2. Conformational distribution of polyubiquitin chains (2).
2. 폐렴구균 유래 감염인자
폐렴구균은 세균성 폐렴을 발생시키는 주된 원인으로, 폐를 비롯한 신체의 다양한 부위에서 심각한 염증을 유발하여 특히 면역력이 약한 환자 및 노인에게는 사망을 초래하는 치명적인 병원체이다. 그럼에도 불구하고, 여러 항생제에 내성을 지닌 다제내성균의 급증과 함께 백신이 모든 유형의 폐렴구균에 유효하지 못한 점 때문에 폐렴구균에 의한 질병의 예방과 치료에 어려움을 겪고 있으며, 전 세계적으로 매년 수많은 희생자를 낳고 있다.
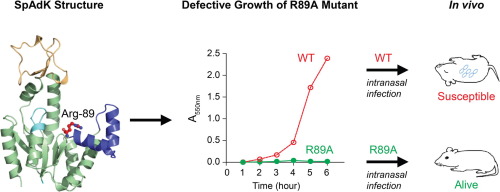
그림 3. 폐렴구균 단백질의 기능 및 구조 연구 (5).
3. 식물 스트레스 대응기작
따라서 스트레스 저항 단백질 간의 상호작용을 연구하고 이것을 사용자 목적에 맞게 조절함으로써 식물의 스트레스 저항성을 향상하여 농업 분야의 생산량 증대에 기여할 수 있을 것으로 기대하고 있다. 하지만 유전자 조작을 통해 유전자재조합작물을 제작하게 되면 섭취 또는 생태계 유해성 검증이 필요할 뿐만 아니라 대중에게 섭취 거부감을 야기하는 문제점이 있다. 따라서, 본 연구실은 유전자재조합작물 제작을 지양하고 생체친화적인 접근 방법으로 ABA 신호 전달 체계 구성요소 간의 상호작용을 조절할 수 있는 펩타이드를 발굴하도록 하여 친환경적인 스트레스 저항 조절이 가능하도록 연구하고 있다.
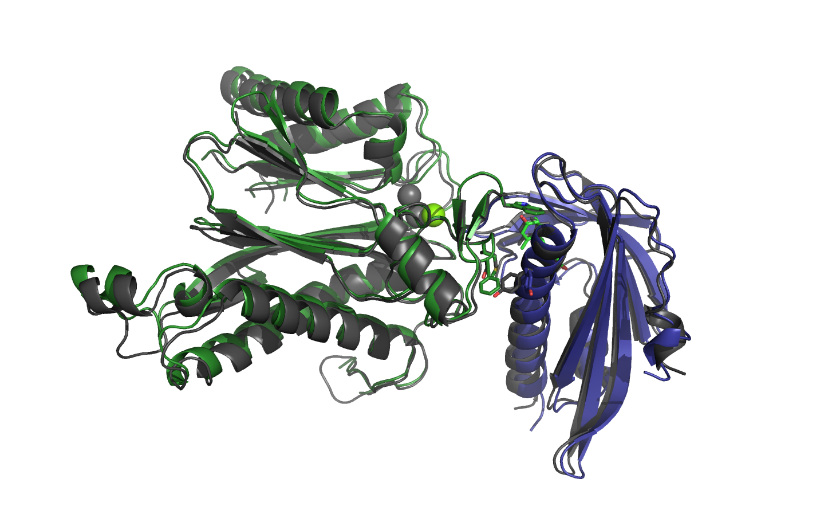
그림 4. 식물의 환경 스트레스 저항 반응 조절을 위한 단백질 엔지니어링
[연구책임자]

이상호 교수
주소: 경기도 수원시 장안구 서부로 2066 성균관대학교 생명과학과 구조생물학 연구실
전화: 031-290-5913
이메일: sangholee@skku.edu
홈페이지: http://sangho.skku.edu
[연구진구성]
지도교수: 이상호
박사후연구원: 신동혁
박사과정:김규희
석∙박사통합과정: 한승수, 이영목
석사과정: 백지석, 김동호
학사과정: 정재은, 박수빈

[대표논문]
1. Jang, S. A., Kim, G. H., Oh, J., Lee, S. Y., Kim, D., Kim, K. H., Kim, Y. H., Rhee, D. K., Lee, S. (2017) Molecular characterization of a single-chain antibody variable fragment (scFv) specific for PspA from Streptococcus pneumoniae. Biochem Biophys Res Commun 482, 141-146.
2. Thach, T. T., Shin, D., Han, S., Lee, S. (2016) New conformations of linear polyubiquitin chains from crystallogrphic and solution-scattering studies expand the conformational space of polyubiquitin. Acta Crystallogr D Struct Biol 72, 524-535.
3. Thach, T. T., Lee, N., Shin, D., Han, S., Kim, G. H., Kim, H., Lee, S. (2015) Molecular determinants of polyubiquitin recognition by continuous ubiquitin-binding domains of Rad18. Biochemistry 54, 2136-2148.
4. Shin, D., Kim, G., Kim, G. H., Xu, Z., Kim, Y. G., Lee, S. (2014) Structural and biochemical characterizations of an intramolecular tandem coiled coil protein. Biochem Biophys Res Commun 455, 339-346.
5. Thach, T. T., Luong, T. T., Lee, S., Rhee, D. K. (2014) Adenylate kinase from Streptococcus pneumoniae is essential for growth through its catalytic activity. FEBS Open Bio 4, 672-682.
6. Han, S., Shin, D., Choi, H., Lee, S. (2014) Molecular determinants of the interaction between Doa1 and Hse1 involved in endosomal sorting. Biochem Biophys Res Commun 446, 352-357.
7. Ta, H. M., Bae, S., Han, S., Song, J., Ahn, T. K., Hohng, S., Lee, S., Kim, K. K. (2013) Structure-based elucidation of the regulatory mechanism for aminopeptidase activity. Acta Crystallogr D Biol Crystallogr 69, 1738-1747.