
생화학분자생물학회입니다.
살아있는 세포의 세포막에서 단일 분자의 확산을 기반으로 한 상피 세포 성장 수용체와 수용성 리간드 간의 상호작용 분석
작성자
김도현, 조카이, 류성호(포항공과대학교)작성일자
2015-08-01조회수
917살아있는 세포의 세포막에서 단일 분자의 확산을 기반으로 한 상피 세포 성장 수용체와 수용성 리간드 간의 상호작용 분석
Analysis of Interactions between the Epidermal Growth Factor Receptor and Soluble Ligands on the Basis of Single-Molecule Diffusivity in the Membrane of Living Cell
Angew Chem Int Ed Engl. 54(24):7028-32, 2015
 |
 |
 |
|
김도현 포항공과대학교 시스템생명공학부 |
조카이 포항공과대학교 생명과학과 |
류성호 포항공과대학교 생명과학과 |
연구배경
리간드-수용체간 상호작용은 세포 안과 밖의 신호전달을 매개함으로써 생명유지에 필수적인 역할을 하는 것 뿐만이 아니라 수많은 질병의 원인 인자로서 현재 개발된 50% 이상의 신약들이 리간드-수용체간 상호작용을 대상으로 하고 있다 (1). 이러한 중요성에도 불구하고 세포막 위에서 일어나고 있는 리간드-수용체간 상호작용에 대한 이해가 많이 부족한 상황이다 (2). 이는 전체 유전자의 15% 이상 되는 다양한 단백질들이 세포막 위에서 매우 높은 농도로 존재함으로써 나타나는 복잡성과 친수성 및 소수성을 모두 가지는 세포막의 생화학적 특이성이 만들어 내는 특수한 환경 때문이다 (3). 따라서 현재까지는 리간드-수용체 간 상호작용의 연구가 시험관 수준에서 대부분 이루어졌으며, 실제 세포막 위에서 나타나는 이들 상호작용의 본질적인 성질을 밝혀내는데 아직까지 기술적으로 큰 한계가 있다.
단백질의 확산을 기반으로 한 영상 기법들을 통해 살아있는 세포에서 단백질들의 고유한 확산 성질을 이용하여 단백질들의 상호작용을 측정할 수 있다 (4). 그러나 세포질의 단백질의 확산과는 달리 수용체의 경우 세포막의 높은 점성도로 인해 확산 성질이 매우 달라진다. 40여 년간 세포막 단백질 유동의 표준모델로서 받아들여져 왔던 샤프만-델브룩 모델에 따르면 세포막의 높은 점성도를 가지는 지질 특성으로 인해 수용성인 리간드가 수용체에 결합하더라도 수용체의 움직임이 변화하지 않는다 (5)(그림 1). 따라서 이러한 가설에 입각하여 수용체의 확산을 기반으로 한 방법들은 다른 방법들에는 없는 많은 장점들에도 불구하고 리간드-수용체 간 상호작용을 측정하는 것이 근본적으로 불가능하다고 생각되어 왔다 (6).
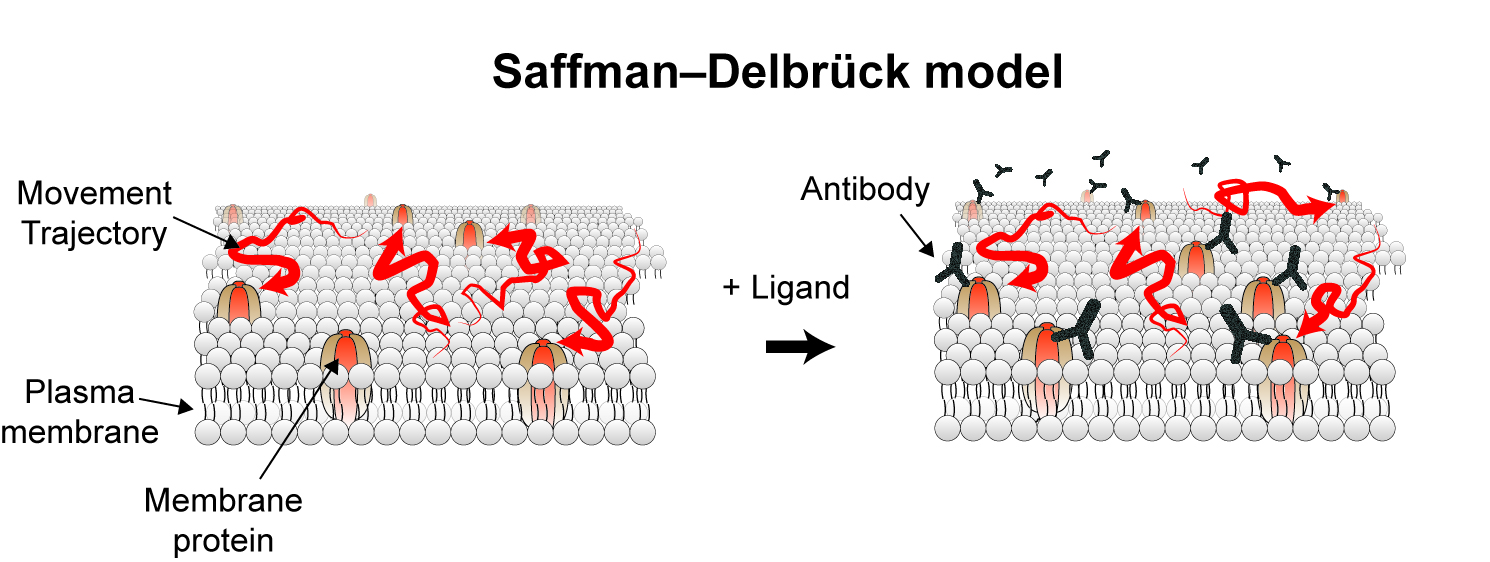
그림 1. 세포 막 단백질 유동의 정설로 받아들여지는 샤프만-델브룩 모델
세포막의 높은 점성도로 인해 막 단백질의 움직임은 Transmembrane Domain에만 의존적이기 때문에 수용성인 리간드의 결합은 막 단백질의 유동에 어떠한 영향도 주지 않음.
연구 결과
최근 초해상도 현미경이 발전하면서 단일 분자 수준에서 수용체의 움직임을 직접 측정할 수 있게 되었다. 살아있는 세포의 세포막 위에서 단일 수용체의 움직임을 단일 분자 추적 기술을 통해 분석하여 각 수용체의 움직임의 속도를 나타내는 확산계수를 결정할 수 있다. 이 방법을 Single Particle Tracking Using Photoactivated Localization Microscopy(sptPALM) 이라고 부른다 (7). 본 연구진은 sptPALM을 통해 살아있는 세포의 세포막 위에서 Epidermal Growth Factor Receptor(EGFR)의 확산을 단일 분자 수준에서 관찰하였고, 우연히 EGFR을 표적으로 하는 항체 항암제인 Cetuximab에 의해 EGFR의 확산이 40% 정도 느려진다는 것을 관찰하였다 (그림 2). 이 관찰을 통해 기존의 샤프만-델브룩 모델과는 달리 리간드가 수용체에 결합하였을 때 수용체의 확산이 변할 수 있을 것이라는 가설을 세우게 되었다.
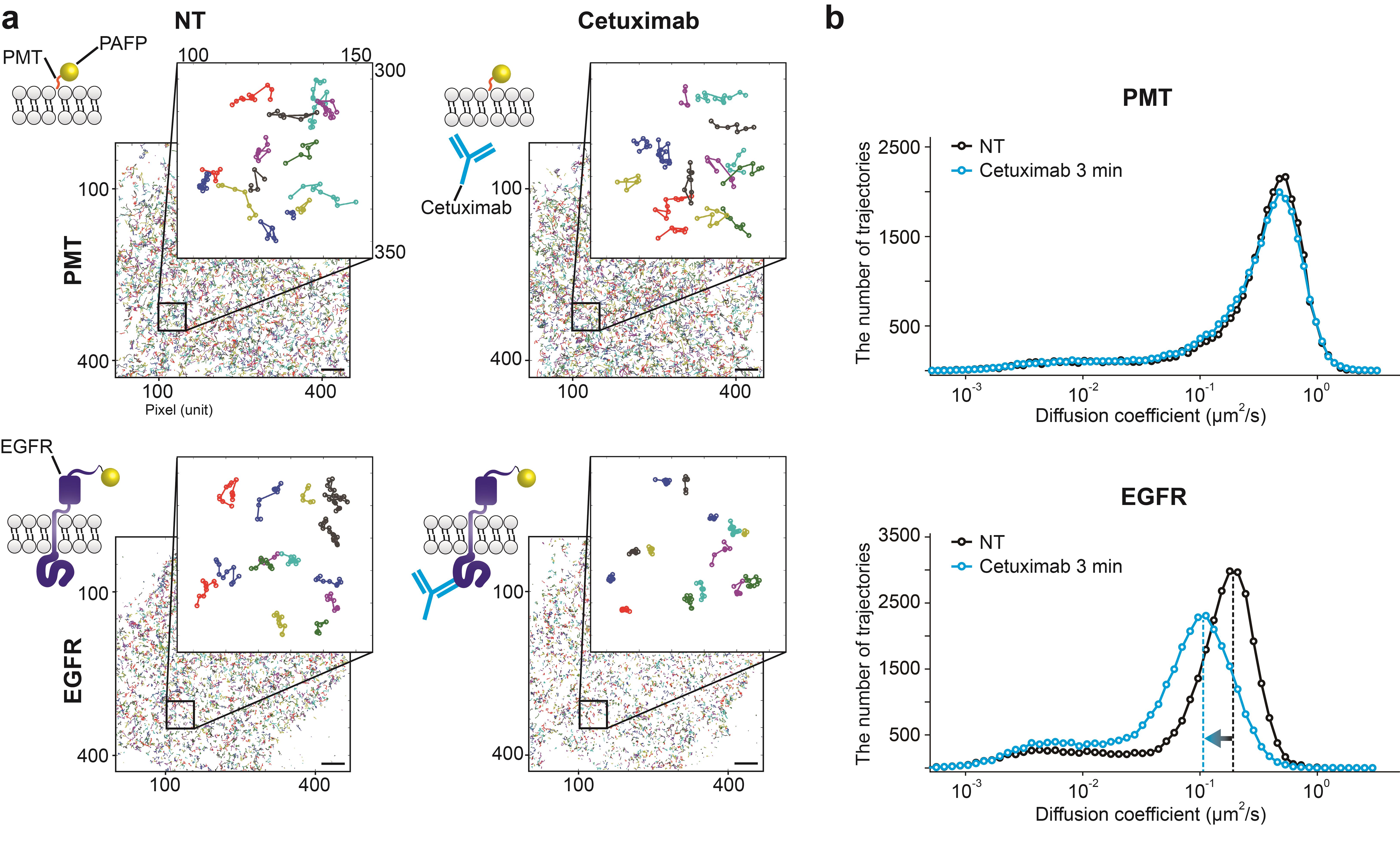
그림 2. Cetuximab에 의한 PMT와 EGFR의 단일분자 수준의 확산의 변화
PMT는 형광단백질을 세포막으로 Localization을 시킨 Negative Control이다. (a) 각 단백질의 단일 분자의 움직임을 보여주는 지도. (b) 각 단백질의 움직임의 확산계수를 보여주는 히스토그램. Cetuximab에 의해 3분 이내에 EGFR 특이적으로 단일 분자 수준에서 확산이 저해되는 것을 알 수 있다.
Cetuximab은 EGFR에 결합하여 Epidermal Growth Factor(EGF)와의 결합을 방해하고 EGFR의 Internalization을 유도한다고 알려져 있다 (8). 따라서 Cetuximab에 의한 EGFR의 확산의 변화는 Cetuximab과 EGFR의 직접적인 결합에 의해 나타나는 것이 아니라 Cetuximab의 이차적인 효과에 의해 나타나는 것일 수 있다. 위의 관찰이 EGF가 없는 환경에서 EGFR의 Internalization이 유도(약 30분 이후)되지 않는 시간인 3분 안에 측정이 이뤄진 것이지만, 이러한 이차적인 효과를 완전히 배제하고 리간드의 직접적인 결합에 의해 EGFR의 확산이 변할 수 있는지 검증하기 위한 실험을 설계하였다. EGFR의 N 말단에 Myc Tag을 Conjugation하였고 EGFR에 어떠한 이차적 효과가 없는 Myc Tag을 표적으로 한 항체를 처리하였을 때 (그림 3a), EGFR의 확산이 Cetuximab과 마찬가지로 40% 정도 저해된다는 것을 관찰할 수 있었다 (그림 3b). 또한 하나의 항체 분자가 두 개의 표적 단백질과 결합할 수 있기 때문에 Myc Tag을 표적으로 하는 항체의 Fab Fragment를 만들어 여전히 EGFR의 확산이 Fab Fragment의 결합에 의해 저해되는 것을 밝혀내었다 (그림 3c). 이 때 EGFR의 확산의 감소 폭이 항체에 비해 조금 줄어든 16% 정도였다. 위의 결과는 Cetuximab 뿐만이 아니라 EGFR의 Extracellular Domain의 서로 다른 Epitope에 결합하는 항체들에 대해서도 같은 결과가 관찰되었다 (그림 3d, e). 또한 EGFR과 같은 Family에 속하는 ErbB2와 ErbB3 수용체들에 대해서도 항체의 직접적인 결합이 이들의 확산을 저해한다는 것을 확인하였다. 흥미롭게도 Single-Transmembrane Domain을 가지는 EGFR Family와 달리 Seven-Transmembrane Domain을 가져 세포막에 매우 의존적인 G-Protein Coupled Receptor(GPCR)에 대해서도 수용성인 항체의 직접적인 결합이 GPCR의 확산을 저해한다는 것을 관찰할 수 있었다. 이 일련의 결과들을 통해 본 연구진은 기존의 사프만-델브룩 모델과 다르게 실제 세포막 위의 수용체의 움직임을 단일 분자 수준에서 관찰하였을 때 리간드의 직접적인 결합이 수용체의 확산을 감소시킨다는 원리를 밝혀내었다. 이 새로운 원리를 바탕으로 리간드-수용체 간 결합을 분석하는 방법을 Single-Molecule Diffusional-Mobility Shift Assay(smDIMSA)라고 명명했다.
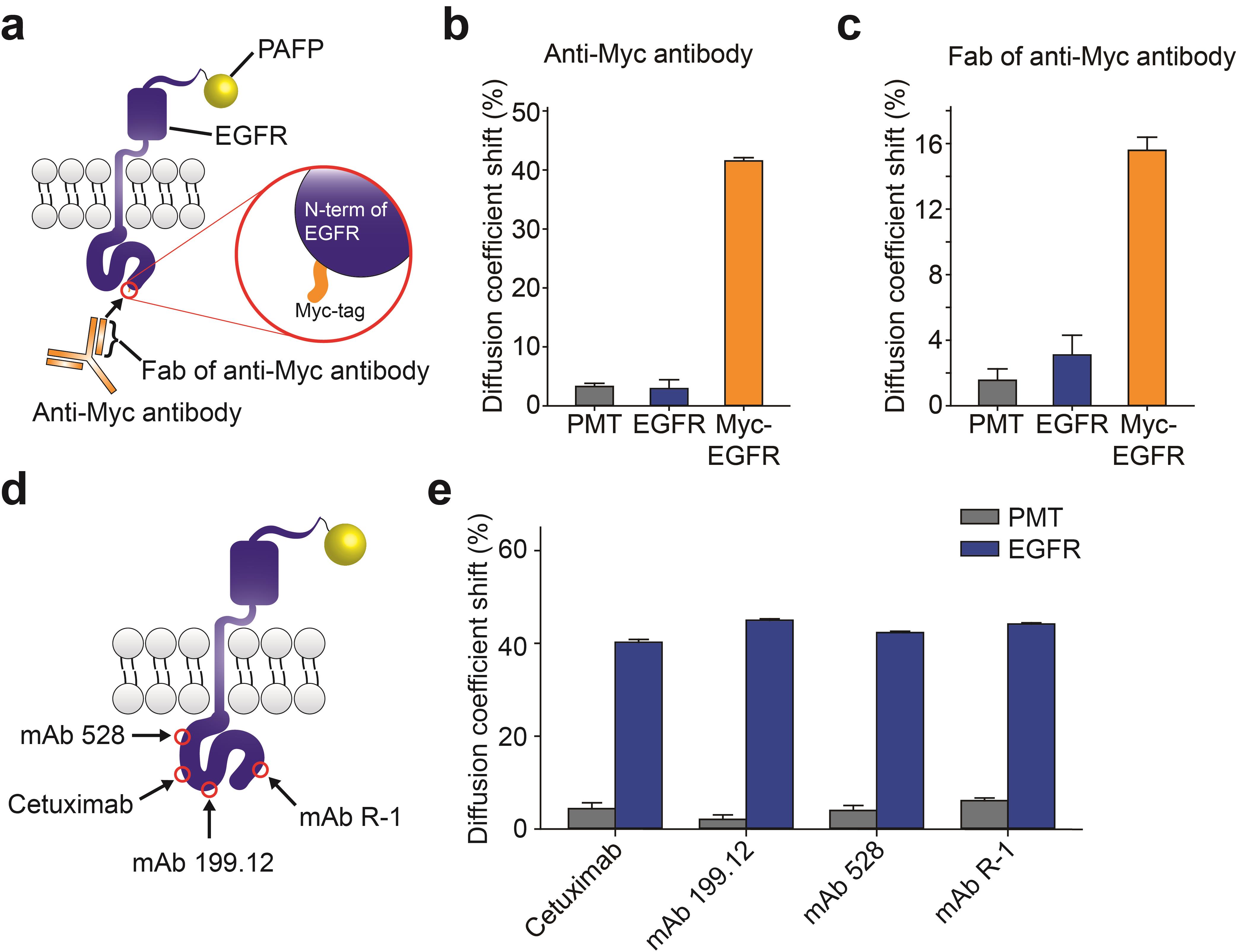
그림 3. 항체에 의한 EGFR 확산 저해의 검증
(a) Myc Tag을 EGFR에 Conjugation한 실험 모식도. (b, c) Myc Tag에 결합하는 항체 (b)와 그 항체의 Fab Fragment (c)에 의한 EGFR의 확산의 변화를 나타낸 그래프. (d) EGFR의 Extracellular Domain의 다른 Epitope에 붙는 항체에 대한 모식도. (e) 각 항체에 대한 EGFR의 확산의 변화를 나타낸 그래프.
smDIMSA는 단일 단백질의 확산을 기반으로하는 방법이기 때문에 리간드가 붙어있는 단백질과 붙어있지 않은 단백질을 단일 분자 수준에서 확산의 차이로 정량적으로 구별할 수 있다. 이러한 성질을 바탕으로 Cetuximab의 농도에 따라 Cetuximab이 EGFR에 붙어있는 정도를 측정함으로써 Cetuximab과 EGFR 간의 해리상수를 살아있는 세포에서 계산할 수 있다. 하나의 COS7 세포에서 Cetuximab의 농도를 점점 증가시켜가며 각 농도에 따른 EGFR의 확산계수를 측정하였고, 이를 통해 Cetuximab의 EGFR에 대한 해리상수를 측정한 결과 약 0.62nM 이었다 (그림 4). 이 결과는 기존의 in vitro에서 측정된 결과와 비슷한 수준이었다. 또한 Cetuximab은 EGFR에 Binding Cooperativity가 없다는 것을 Scatchard Plot을 통해 알 수 있었다.
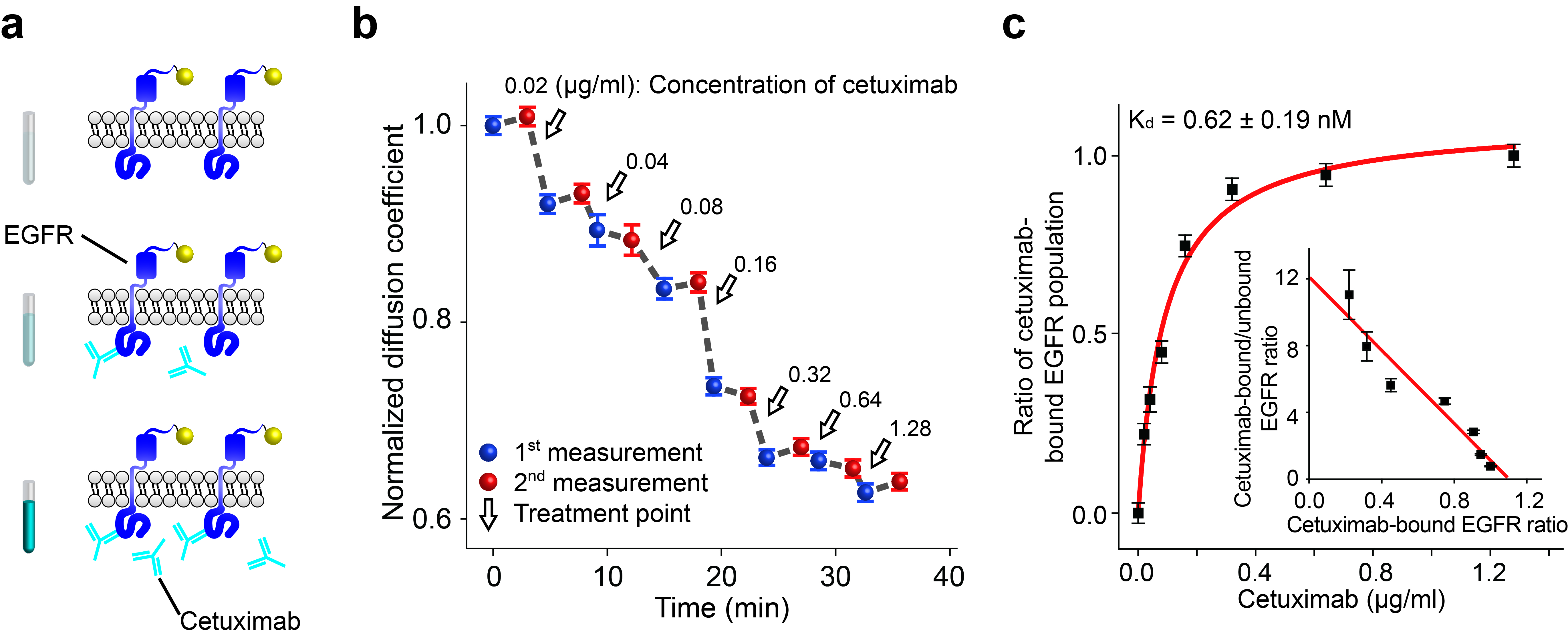
그림 4. 리간드 수용체 간 상호작용의 정량적인 분석
(a) Cetuximab의 농도에 따른 EGFR과의 결합 정도에 대한 단분자 수준에서의 모식도. (b) 단일 세포에서 Cetuximab의 농도에 따른 EGFR의 확산계수의 변화. (c) Cetuximab과 결합한 EGFR과 결합하지 않은 EGFR의 확산의 차이를 통한 해리상수의 측정 및 Scatchard Plot을 통한 Cooperativity의 분석.
흥미롭게도 여러 종류의 암에서 많이 발견되는 EGFR의 Mutant인 EGFR L858R에 대한 Cetuximab의 결합을 정량적으로 분석하였을 때, Cooperativity가 없었던 Wild-Type과는 달리 Positive Cooperativity가 나타나는 것을 관찰하였다 (그림 5a). Cooperativity는 두 개 이상의 결합 위치가 존재할 때 하나의 위치에 대한 결합이 단백질의 Conformational Change를 유도하여 다른 위치의 결합에 대한 Affinity를 변화시킬 때 나타난다. EGFR L858R은 보통 Monomer로 존재하는 Wild-Type EGFR과 달리 대부분 Dimer 상태로 존재한다고 알려져 있다 (9). 따라서 이 Positivie Cooperativity는 첫번째 Cetuximab 분자가 Dimerized EGFR L858R과 결합할 때는 Unfavorable 하지만, 이 결합이 Dimerized EGFR L858R의 Conformational Change를 일으켜 두번째 Cetuximab 분자가 결합할 때는 Facilitated 되는 것이라고 해석할 수 있다. Cetuximab은 구조적으로 EGFR의 Extracellular Domain이 Tethered Form일 때 결합을 할 수 있다. 따라서 Dimerized EGFR L858R의 Extracellular Domain의 Conformation은 Cetuximab이 결합하기 어려운 Extended Form이라는 것을 유추해내었다. 이를 뒷받침하기 위해 EGFR의 Extended Form에도 결합할 수 있는 항체인 mAb 199.12와 EGFR L858R 간의 결합을 정량적으로 분석하였을 때 어떠한 Cooperativity도 나타나지 않는 것을 확인할 수 있었다 (그림 5b). 또한 첫번째 Cetuximab 분자가 Dimerized EGFR L858R에 결합하였을 때, 결합한 쪽의 EGFR의 Extracellular Domain이 Tethered Form이 되면서 나머지 Cetuximab이 결합하지 않은 EGFR의 Extracellular Domain도 Extended Form에서 Favorable한 Tethered Form으로 전환되어야 Positive Cooperativity가 나타날 수 있다. 이것은 기존에 잘 알려진 EGFR의 Extracellular Domain의 Dimerization Arm 간의 상호작용 때문일 것이라 추측하였다. 따라서 EGFR L858R과 같이 Dimer 상태로 존재하지만 Dimerization Arm이 Deletion 되어있는 Mutant인 EGFRvIII에 대한 Cetuximab의 결합을 정량적으로 분석하였고, 그 결과 어떠한 Cooperativity도 나타나지 않는다는 것을 알 수 있었다 (그림 5c). 이 일련의 결과를 통해 기존에 Controversial 했던 Dimerized EGFR L858R의 Extracellular Domain의 Conformation을 밝혀내었다 (그림 5d).
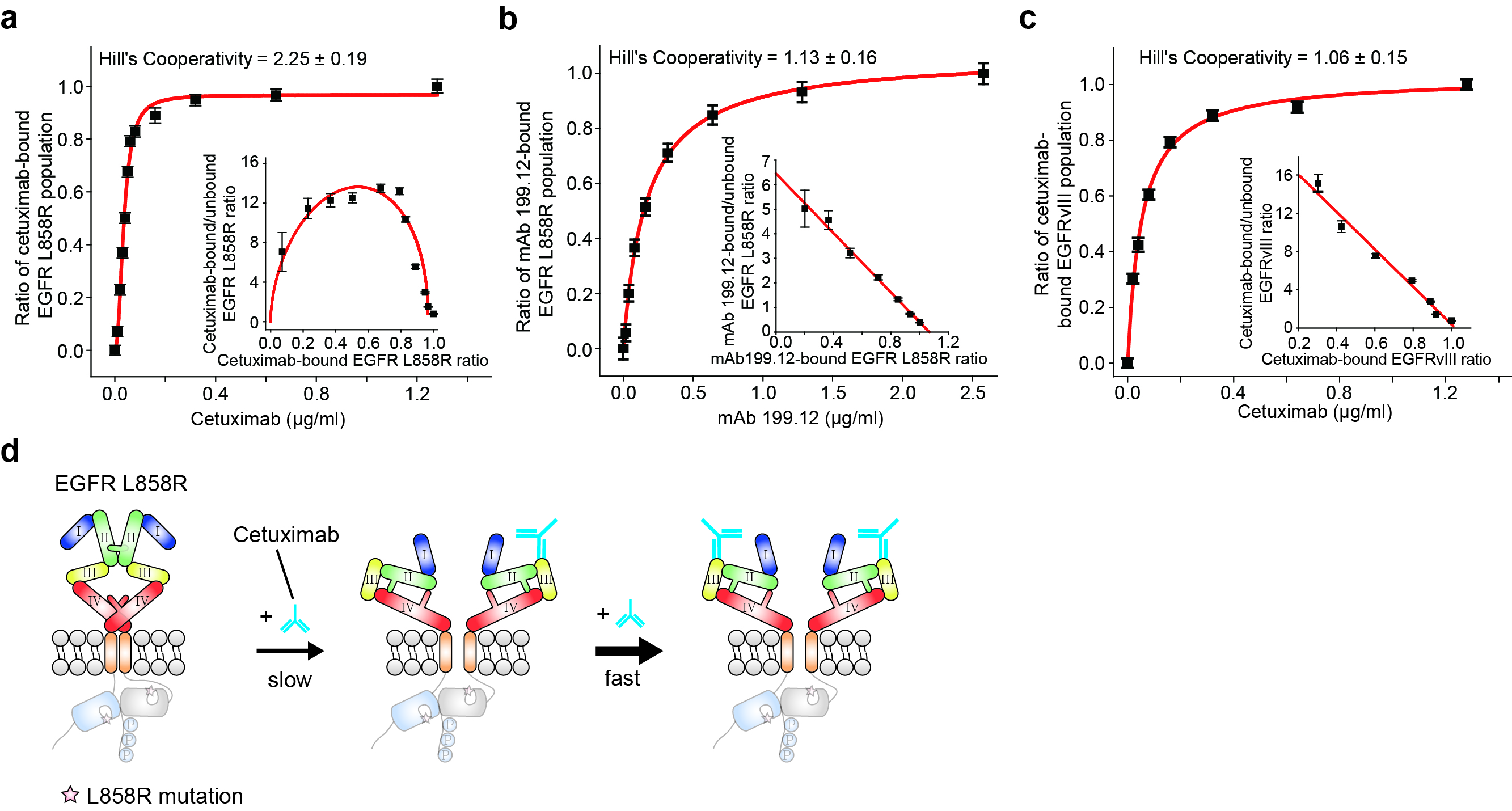
그림 5. EGFR L858R에 대한 Cetuximab 결합의 Positive Cooperativity
(a, b) EGFR L858R에 대한 Cetuximab (a)과 mAb 199.12 (b)의 해리상수 및 cooperativity의 분석. (c) EGFRvIII에 대한 Cetuximab의 해리상수 및 Cooperativity의 분석. (d) EGFR L858R에 대한 Cetuximab의 Positive Cooperativity를 설명하는 모델.
연구의 성과 및 의의
40여 년간 세포막 단백질 유동의 표준모델로서 받아들여져 왔던 1975년에 정립된 샤프만-델브룩 모델에 따르면 인지질 이중층으로 이루어진 세포막의 높은 점성도로 인해 수용성인 리간드가 수용체에 결합하더라도 수용체의 움직임이 변화하지 않는다고 믿어져왔다. 그러나 본 연구에서 기존에 측정이 불가능했던 단일 분자 수준에서의 세포막 수용체의 움직임을 직접적으로 관찰함으로써 리간드가 수용체에 결합하였을 때 그 움직임이 변화한다는 사실을 밝혀냄으로써 막 단백질의 유동 이론에 대한 기존의 패러다임을 바꿀 수 있는 매우 중요한 증거를 제시하였다.
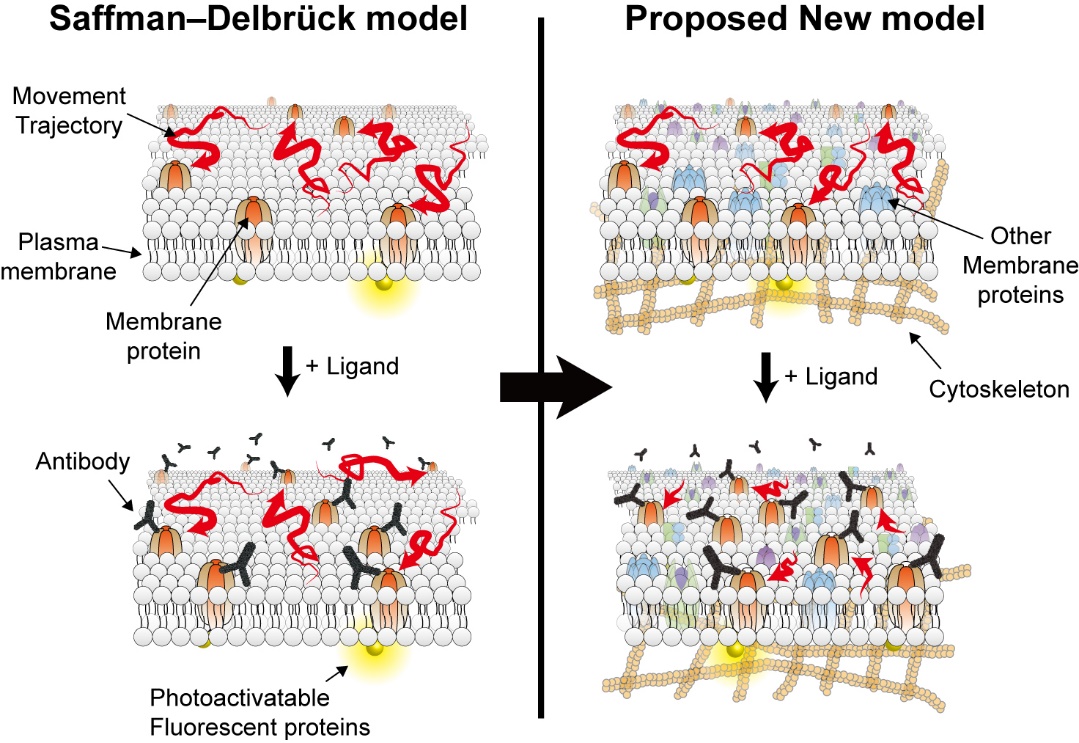
그림 6. 세포막 유동에 대한 기존 모델과 새롭게 제안된 모델의 차이점
인지질 이중층으로 이루어진 세포막에 박혀서 움직이고 있는 막 단백질들의 단일분자 수준에서의 움직임을 자외선을 쐬게 되면 형광성질을 나타내는 광활성 형광단백질을 이용함으로써 관찰할 수 있다. 40년 동안 정설로 받아드려져 왔던 샤프만-델브룩 모델에 따르면 세포막의 높은 점성도로 인해 막 단백질의 움직임이 제한되어 수용성인 리간드가 수용체에 결합하더라도 그 움직임이 변화되지 않는다 (왼쪽). 그러나 실제 세포막 위에서 막 단백질의 움직임을 직접적으로 관찰한 결과 리간드가 수용체에 결합하였을 때 그 움직임이 둔화된다는 것을 밝혀내었다. 이 현상은 기존모델에서 고려하지 않았던 살아있는 세포의 세포막 위에서 수많은 종류의 막 단백질들이 매우 복잡하게 동시다발적으로 움직이고 있다는 점과 세포 골격으로 인해 막 단백질의 움직임이 제한된다는 점 때문에 나타난다고 예측되고 있다 (오른쪽).
본 연구에서 개발된 기술인 smDIMSA는 수용체에 결합하는 리간드의 크기를 정량적으로 인식하는 방법을 사용하기 때문에 리간드에 어떠한 표지 없이 대상 수용체에 대한 신약 리간드의 스크리닝이 가능하며, 기존의 방법들과 비교하였을 때 혁명적인 장점들을 가지고 있다. 리간드-수용체의 결합을 측정하기 위해서는 일반적으로 리간드에 방사성 동위 원소나 형광물질을 화학적으로 표지해야 검출이 가능하다. 그러나 수많은 종류의 신약 리간드 후보들을 선별하기 위해서는 이러한 표지방법은 엄청난 비용과 시간이 필요하기 때문에 신약 선별을 위해서는 리간드 비표지적 방법이 필수적이다. 현재 상용화되어 주로 사용되고 있는 표면 플라즈몬 공명(Surface Plasmon Resonance)을 이용한 비표지적 방법은 복잡하고 힘들게 정제한 대상 단백질을 값비싼 금표면에 부착시켜 리간드와의 결합을 측정하게 된다 (10). 특히 수용체의 경우 높은 세포막 의존도로 인해 정제가 어려워 그 측정이 더욱 힘들었다. smDIMSA는 살아있는 세포에서 리간드-수용체간 상호작용을 직접 측정함으로써 시험관 실험(in vitro Assay)에서 오는 양성 및 음성 오류들을 현저히 줄일 수 있고 까다로운 세포막 단백질 정제 과정을 완전히 없애고 비용적인 측면에서 기존대비 수천 배 이상 절약할 수 있다. 따라서 본 연구는 신약 후보 물질을 선별하는 데도 큰 기여를 할 것이라고 기대하고 있다.
참고문헌
1. Jennings, M.L. (1989) Topography of membrane proteins. Annu Rev Biochem 58, 999-1027.
2. Babu, M., Vlasblom, J., Pu, S., Guo, X., Graham, C., Bean, B.D., Burston, H.E., Vizeacoumar, F.J., Snider, J., Phanse, S., Fong, V., Tam, Y.Y., Davey, M., Hnatshak, O., Bajaj, N., Chandran, S., Punna, T., Christopolous, C., Wong, V., Yu, A., Zhong, G., Li, J., Stagljar, I., Conibear, E., Wodak, S.J., Emili, A., Greenblatt, J.F. (2012) Interaction landscape of membrane-protein complexes in Saccharomyces cerevisiae. Nature 489, 585-589.
3. Jacobson, K., Sheets, E.D., Simson, R. (1995) Revisiting the fluid mosaic model of membranes. Science 268, 1441-1442.
4. He, H., Marguet, D. (2011) Detecting nanodomains in living cell membrane by fluorescence correlation spectroscopy. Annu Rev Phys Chem 62, 417-436.
5. Saffman, P.G., Delbruck, M. (1975) Brownian motion in biological membranes. Proc Natl Acad Sci USA 72, 3111-3113.
6. Lippincott-Schwartz, J., Snapp, E., Kenworthy, A. (2001) Studying protein dynamics in living cells. Nat Rev Mol Cell Biol 2, 444-456.
7. Manley, S., Gillette, J., Patterson, G., Shroff, H., Hess, H., Betzig, E., Lippincott-Schwartz, J. (2008) High-density mapping of single-molecule trajectories with photoactivated localization microscopy. Nat Methods 5, 155-157.
8. Perez-Torres, M., Guix, M., Gonzalez, A., Arteaga, C.L. (2006) Epidermal growth factor receptor (EGFR) antibody down-regulate mutant receptors and inhibits tumors expressing EGFR mutants. J Biol Chem 281, 40183-40192.
9. Shan, Y., Eastwood, M.P., Zhang, X., Kim, E.T., Arkhipov, A., Dror, R.O., Jumper, J., Kuriyan, J., Shaw, D.E. (2012) Oncogenic mutations counteract intrinsic disorder in the EGFR kinase and promote receptor dimerization. Cell 149, 860-870.
10. Maynard, J.A., Lindquist, N.C., Sutherland, J.N., Lesuffleur, A., Warrington, A.E., Rodriguez, M., Oh, S.H. (2009) Surface plasmon resonance for high-throughput ligand screening of membrane-bound proteins. Biotechnol J 4, 1542-1558.
첨부파일