
생화학분자생물학회입니다.
폐 선암을 유발하는 유전체 구조 변이의 발생기전 연구
작성자
관리자작성일자
2019-08-01조회수
1169폐 선암을 유발하는 유전체 구조 변이의 발생기전 연구
Tracing oncogene rearrangements in the mutational history of lung adenocarcinoma
Cell 177(7): 1842-1857, 2019
| | | |
이준구 KAIST 의과학대학원 (현 Harvard Medical School) soarineagle82@gmail.com | 박성열 의과학대학원 syparkmd@kaist.ac.kr | 주영석 KAIST 의과학대학원 ysju@kaist.ac.kr | 김영태 서울대학교 의과대학 ytkim@snu.ac.kr |
연구배경
사람의 정상 세포는 어떻게 암세포로 돌변하는가? 이것은 종양학의 오래 된 질문이다. 약 30여년 전 유전자의 단일 돌연변이가 정상세포를 암세포로 변화시키는 데 충분하다는 기념비적인 발견 이후 (1) 발암의 원인 돌연변이 및 암유전자의 체계적인 발굴은 암유전체 연구의 중요한 목표였다 (2). 특히 지난 10년 간 초고속 유전체 서열 기술 (Next Generation Sequencing)을 이용하여 수많은 연구가 진행된 바 있다. 하지만 높은 비용 및 분석 기술의 현실적인 한계로 인하여 대부분의 연구는 전장 유전체 (Whole-Genome) 대신 약 2만개 정도의 protein-coding 유전자의 엑손 영역을 중심으로 (Whole-Exome Sequencing) 진행되었으며, non-coding 영역에서 주로 일어나는 유전체의 rearrangement, 즉 유전체 구조 변이 (Structural Variation; SV)에 대한 이해는 매우 부족한 실정이다. 하지만 일부 암들은 명백하게 구조변이에 의해 발생한다. 예를 들어 약 10% 정도의 폐 선암(Lung Adenocarcinoma)은 KRAS나 EGFR 유전자의 point mutation이 아니라, ALK, RET, ROS1등 protein tyrosine kinase 유전자들이 유전체 상에서 멀리 떨어진 다른 파트너 유전자와의 융합에 의해 발생한다 (3). 이러한 융합 유전자가 생성되기 위해서는, 유전체의 역위 (Inversion), 전좌 (Translocation), 결실 (Large-Deletion)등의 SV가 수반되어야 한다. 비록 이전까지 많이 알려진 사실은 없었지만, 우리 그룹의 이전 연구 결과는 chromothripsis 나 chromoplexy 등 (후술함) 복잡구조변이 (Complex Genome Rearrangement) 기전이 고형암에서 융합 유전자를 만드는 일반적인 기전일 수 있음을 제시하고 있었다 (4, 5). 이에 우리는 whole-genome sequencing (WGS)을 이용하여 융합유전자에 의해 발생한 폐 선암의 유전체 서열을 규명하고자 하였다. 본 글에서는 이번 논문에서 새로이 밝힌 내용 가운데 핵심 내용을 추려 정리하였다.
연구결과
1. 암유전체 데이터 생성
우리는 총 138 례의 폐 선암 WGS data를 bioinformatics를 통해 다각도로 정밀 분석하였다. 이 중 49 례는 이번 연구를 위하여 새로 시퀀싱을 수행하였고 나머지는 기존에 시퀀싱 된 서열을 새로운 각도로 분석하였다. 융합유전자에 의한 폐 선암을 많이 포함시키기 위해 (일반적인 빈도는 약 10%임) 약 350례의 환자 유래 폐 선암 조직을 새로 screening하여 그 가운데 융합유전자를 가진 케이스를 선정하였으며, 결과적으로 본 WGS 코호트의 약 28%인 39 케이스가 융합유전자를 포함하고 있었다. 암 세포 특이적 돌연변이 (Somatic Mutation)를 효과적으로 찾아내기 위하여 같은 환자의 정상 조직 (혈액)을 함께 시퀀싱하여 암조직의 서열과 비교하였다. 전체적으로 정상 조직은 약 30x, 암 조직은 약 40x로 시퀀싱 하였다 (이는 구조변이를 민감하고 정밀하게 발굴할 수 있는 최소수준의 데이터 양에 해당한다) 이들 일차 서열 데이터로부터 WGS로부터 찾아 낼 수 있는 모든 종류의 somatic mutation을 (SNV, Short-Indel, SV, CNV, Mobile Element 등) 정밀 발굴하였다. 그리고 이들 돌연변이들을 이용하여, 1) 암세포에서 이들 돌연변이가 발생하는 분자적인 기전을 이해하고 2) 돌연변이를 하나의 마커로 이용하여 암 발생 과정을 재구성 (Reconstruction)하는 유전체 해석에 (Interpretation) 상당한 시간 및 노력을 기울였다.
2. 폐 선암에서 융합유전자는 복잡 유전체 구조 변이에 의해 생성된다
본 연구의 핵심 발견 가운데 하나는 다수의 융합유전자의 발생이 large-deletion/inversion/translocation 등의 단순 구조변이 (Simple Genome Rearrangement)가 아니라 복잡구조변이에 (Complex Genome Rearrangement) 의해 일어난다는 사실이다. 예를 들어 Fig. 1a 와 같이 융합유전자를 일으키는 두 유전자가 한 염색체에서 반대 방향의 orientation으로 위치한 경우, 융합유전자를 생성시키는 가장 단순한 유전체 변이는 해당 염색체에 2개의 double-strand break (DSB)가 생긴 후 유전체 조각의 역위 (Inversion)가 일어나는 것이다 (Simple Rearrangement). 하지만 실제로 폐 선암에서는, 염색체가 광범위하게 부서지고 재조합되는 과정 가운데에서 대다수 (~75%) 융합 유전자가 생성되고 있었다 (Complex Rearrangement) (Fig 1). 복잡구조변이에는 실제 여러 다른 기전들이 있지만, 2010년대 초반 발견된 chromothripsis나 chromplexy가 대표적이다 (5-7).
일반적으로 복잡구조변이 과정에서는 부서진 많은 유전체 조각들의 결실 (Copy Number Loss)이 빈번히 발생한다고 알려져 있었으나, 이번 연구에서 융합유전자를 형성하는 복잡구조변이에서는 결실의 빈도가 상대적으로 매우 낮은 특성을 보였다 (Copy Number Balanced). 이러한 특성은 융합유전자들이 세포가 유전체 불안정성 (Genome Instability)을 확보하기 전, 즉 암세포의 특성을 얻은 시점보다 훨씬 이전, ‘정상세포’ 시기에 발생하였다는 사실을 시사하고 있다.
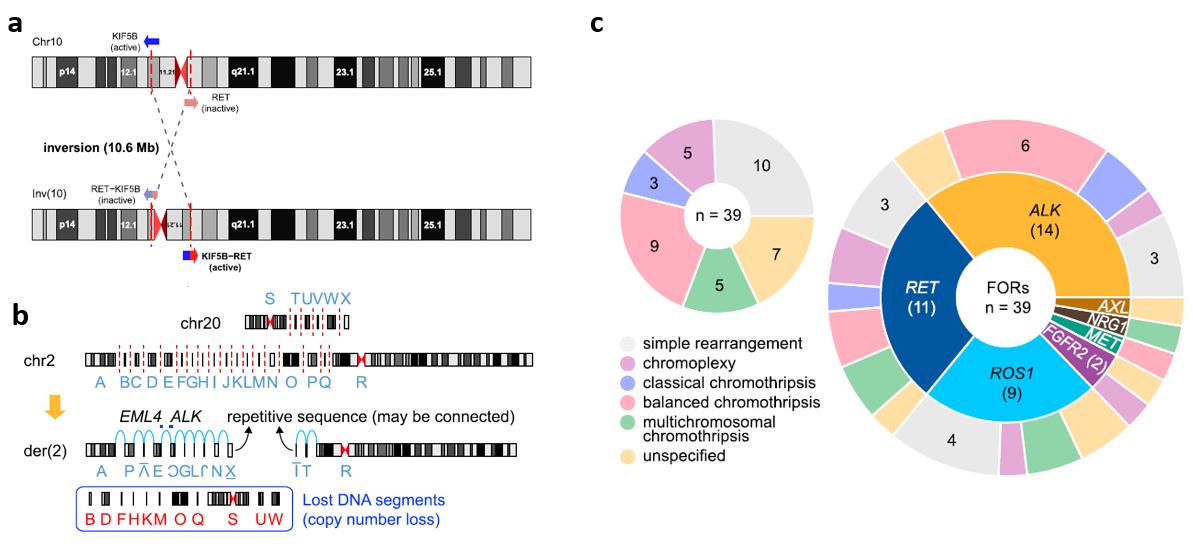
Fig 1. 복합유전체 구조변이에 의한 폐 선암을 유발하는 융합유전자의 생성. (a) balanced inversion에 의한 KIF5B-RET 융합유전자 생성 (b) 복잡구조변이의 예 (Chromothripsis). 2번 염색체가 약 20개 가까운 fragment로 쪼개지는 DSB crisis 후 재조합되는 과정에서 융합유전자가 발생한다 (c) 대다수의 (~75%) 융합유전자는 complex rearrangement에 의하여 생성됨.
3. 융합유전자는 매우 어린 나이에 생성될 수 있다
우리는 융합유전자의 발생 시점을 ‘mutation-time analysis’를 이용하여 체계적으로 추정하였다. 현재 몇 그룹에 의해서 시도되고 있는 이 분석 방법의 이론적 배경은 사실 매우 단순하다 (8). 암세포에서는 빈번하게 유전체 조각들의 증폭 (Copy Number Gain)이 발생하는데, 증폭 시점 이전에 해당 유전체 조각에 쌓인 point mutation들은 amplicon의 모든 copy에 함께 존재하게 되고 (co-amplification이 일어남), 증폭 시점 이후에 해당 유전체 조각에 쌓인 point mutation들은 함께 증폭될 수 없다. 덧붙여 유전체에 쌓이는 내재적 point mutation들은 시간에 비례하여 누적되는 ‘clock-like property’를 가지고 있으므로 (9), copy number gain지역의 point mutation들의 양상을 분자 시계로 (Molecular Clock) 이용하면 증폭이 일어난 시점을 대략적으로 추정할 수 있게 된다.
우리가 연구한 융합유전자 가운데 상당수는 해당 지역의 local copy number gain과 함께 co-amplification되어 있었는데, 1) 이는 융합유전자들의 생성 시점이 copy number gain이 일어난 시점 이전이라는 것을 시사한다. 반면 copy number gain과 함께 co-amplification이 일어난 point mutation의 빈도는 상대적으로 매우 낮았는데, 2) 이는 증폭이 일어난 시점이 환자의 인생에서 매우 일러 충분한 양의 point-mutation이 쌓일 수 있는 시간이 부족한 시나리오에 부합한다. 1) 및 2)의 관찰을 융합하면, 우리는 융합유전자의 생성이 환자의 일생에서 매우 이른 시기에 일어났다는 결론에 필연적으로 도달하게 된다. 정밀한 통계적 접근을 적용하면 평균적으로 융합유전자는 암 진단 시점으로부터 약 30년 전, 즉 20대 정도에 발생하는 것으로 추정되었으며 10대 이전 유년기에 융합유전자가 생성된 것으로 보이는 극단적인 케이스도 있었다 (2). 결론적으로 우리의 연구 결과는, 염색체가 광범위하게 파괴되었다가 재조합되는 복잡구조변이 현상이 인생의 매우 이른 시기에 폐 상피 세포들에서 일어날 수 있으며, 이러한 변이를 가진 세포는 수 십년의 잠복기 후에 폐암세포로 진화할 수 있다는 사실을 보여주고 있다 (Fig. 3).
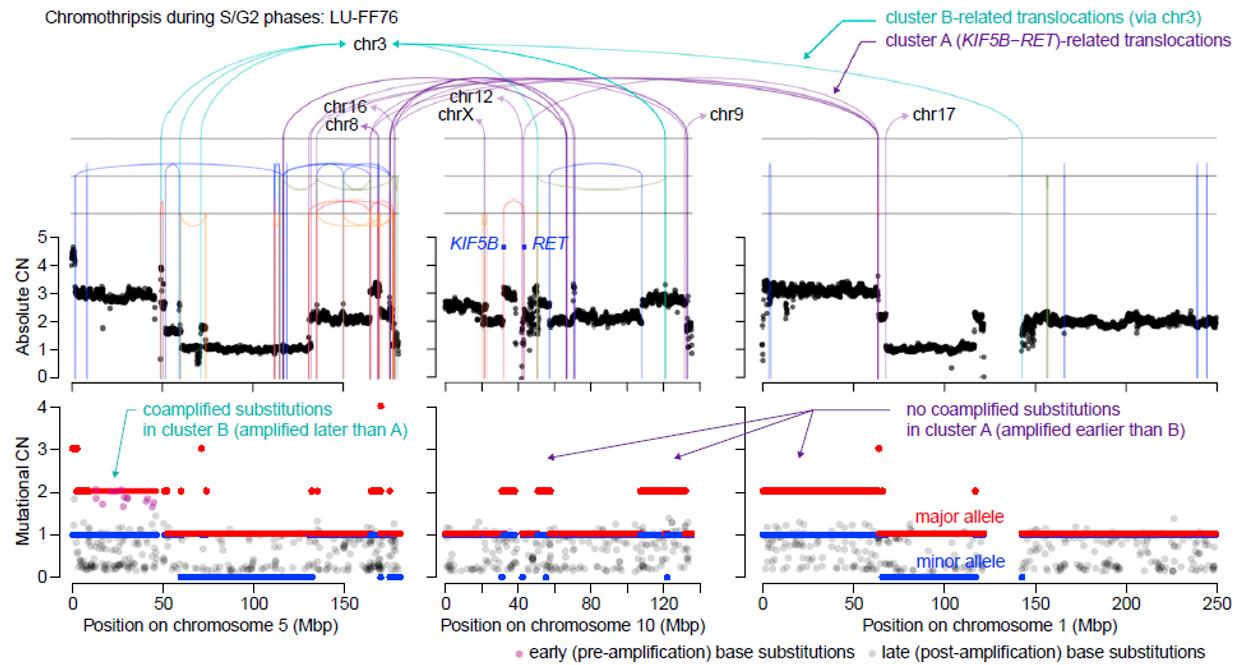
Fig 2. 융합유전자가 매우 어린 나이에 발생할 수 있음을 보여주는 예 (Case LU-FF76).
KIF5B-RET 융합유전자의 증폭이 일어났지만 co-amplification 된 point-mutation의 수는 없어, 매우 유년기에 융합유전자가 생성되었음을 시사함.
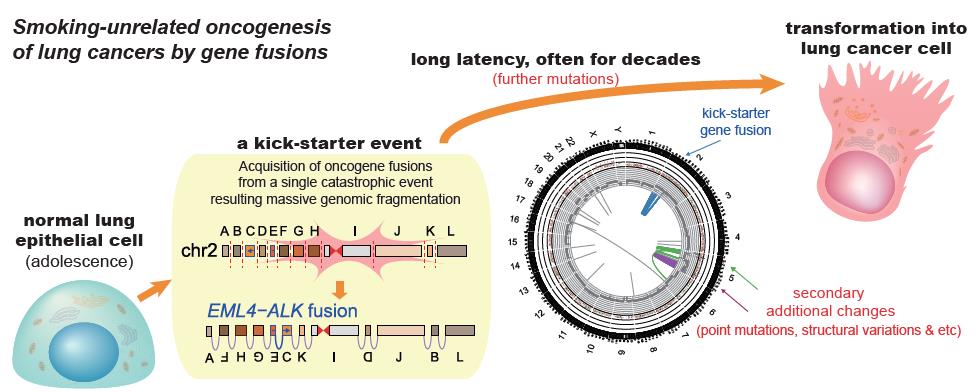
Fig 3. 융합유전자에 의한 폐 선암의 생성과정 모델
4. 융합유전자에 의한 폐암은 일반적인 폐암과 다르다
일반적으로 폐 선암의 가장 큰 원인 인자는 흡연으로 알려져 있다. 담배 연기에 포함된 benzopyrene과 같은 carcinogen은 이에 노출된 폐 상피세포의 DNA에 손상을 입히며, 결과적으로 주로 C:G>A:T 돌연변이를 생성하게 된다 (10; ‘Signature 4 돌연변이’로 불림). 따라서 흡연자의 폐 선암 유전체는 비흡연자 폐암 유전체에 비해 압도적으로 많은 Signature 4 돌연변이가 존재한다. 반대로 암세포에서 보이는 Signature 4 돌연변이의 빈도를 이용하면 그 암세포가 흡연에 노출된 정도를 추정할 수도 있다. 흥미롭게도 모든 융합유전자에 의한 폐 선암 유전체에서는 Signature 4 돌연변이의 빈도가 매우 낮았으며, 이는 융합유전자의 생성이 흡연과는 큰 관련이 없음을 강력히 시사한다. 일반적인 폐 선암들의 유전체 변이 양상과는 다르게 융합유전자에 의한 폐 선암은 1) point mutation의 개수가 (Tumor Mutational Burden) 흡연에 의한 폐 선암에 비하여 약 10% 내외로 매우 낮았으며 2) 구조 변이의 빈도 역시 상대적으로 낮아서, 전체적으로 유전체 돌연변이의 수가 매우 적은 특성을 보였다. 뿐만 아니라 일반적으로 폐 선암에서 50-60%의 빈도로 보이는 3) TP53 (p53) 유전자 돌연변이도 20% 내외로 매우 낮았다. 반면 4) SETD2 유전자의 truncation mutation의 비율은 약 20%로서 일반 폐 선암에 비하여 매우 높았다 (5배 이상) (Fig. 4). 이는 SETD2 유전자의 불활성화가 융합유전자에 의한 암 발현과 밀접한 관련이 있다는 점을 시사하고 있다. 결론적으로 융합유전자에 의한 폐 선암은 흡연에 의한 폐 선암과는 발생과정 뿐만 아니라 유전체 돌연변이 특성이 매우 다르다는 점을 관찰할 수 있었다.
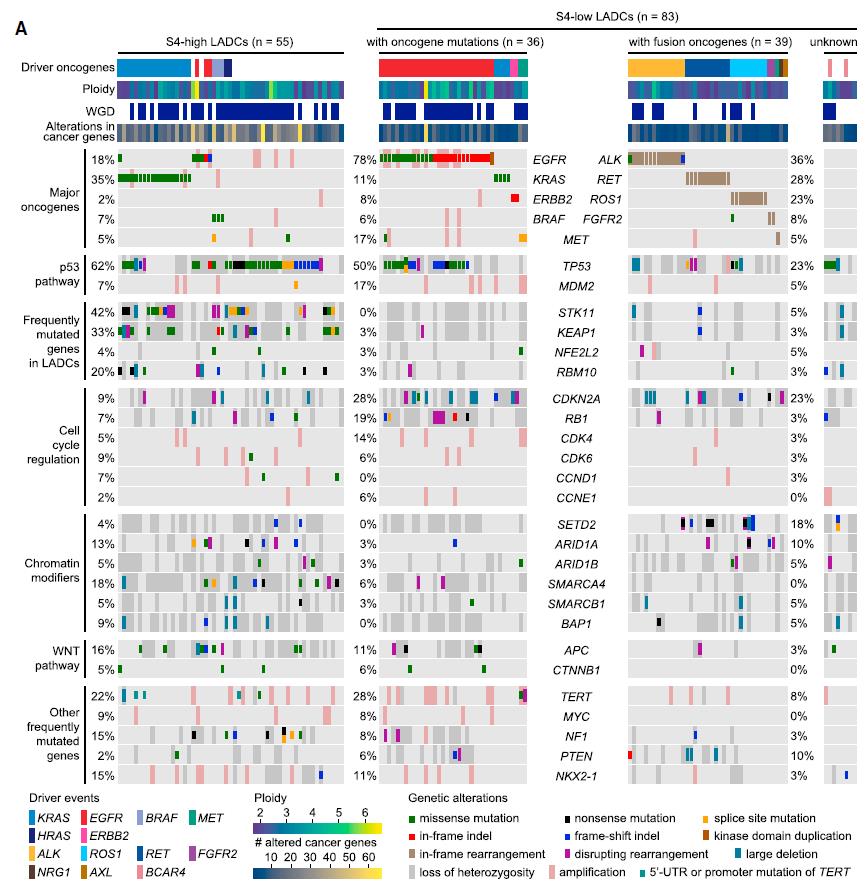
Fig 4. 138례 폐 선암의 mutational landscape. 각 케이스는 (1) 흡연에 의한 폐암 (맨 왼쪽 column), (2) 비흡연자-point mutation에 의한 폐암 (왼쪽 두번째 column), (3) 비흡연자-융합유전자에 의한 폐암 (왼쪽 세번째 column), (4) 비흡연자-돌연변이 미확인 폐암 (맨 우측 column)의 네 그룹으로 분류하였음.
연구의 성과 및 의의
본 연구는 폐 선암의 전장 유전체를 체계적으로 분석한 첫 번째 연구 결과이다. 본 연구자들은 2012년 폐 선암의 원인 융합유전자인 KIF5B-RET를 RNA sequencing등을 이용해 처음으로 발견해 보고한 바 있다 (11). 하지만 이러한 융합유전자들이 실제로 폐 상피세포에서 어떻게 발생하는 지는 미스테리였으며, 이번 연구를 통해 체계적 정리가 시작된 셈이다. 하지만 우리의 연구 결과는 다시 수 많은 새로운 질문을 던지고 있다. 그 가운데 하나는 사람의 ‘정상 세포에서 복잡구조변이 기전을 유발시키는 원인은 과연 무엇인가’ 일 것이다. 이를 알아낼 수 있다면, 융합 유전자에 의한 폐 선암을 예방하거나 조기발견 할 수 있는 길이 열리기 때문이다. 실험적으로는 chromosomal segregation error (lagging-chromosome에 의한 micronucleus) 나 chromosome end-to-end fusion에 의한 anaphase bridge 형성이 하나의 가능성으로 제시된 바 있다 (12,13). 이러한 원인들이 실제로 우리 몸에서 일어나고 있는지, 또 다른 원인들은 무엇이 있는지 체계적으로 규명하는 것이 바로 다음 연구의 중요한 목표가 될 것이다.
참고문헌
1. Tabin, C.J. et al. (1982) Mechanism of activation of a human oncogene. Nature 300(5888):143-9.
2. Stratton, M.R. et al. (2009)The cancer genome. Nature 458(7239):719-24.
3. Herbst, R.S. et al. (2018) The biology and management of non-small cell lung
cancer. Nature 553(7689):446-454
4. Lee, J.K. et al. (2017) Complex chromosomal rearrangements by single catastrophic pathogenesis
in NUT midline carcinoma. Annals of Oncology 28(4):890-897.
5. Yi, K. et al (2018) Patterns and mechanisms of structural variations in human cancer.
Experimental and Molecular
Medicine 50(8):98.
6. Stephens, P.J. et al. (2011) Massive genomic rearrangement acquired in a single catastrophic event during cancer
development. Cell 144(1):27-40.
7. Baca, S.C. et al. (2013) Punctuated evolution of prostate cancer genomes. Cell 153(3):666-7.
8. Gerstung, M. et al. (2017) The evolutionary history of 2,658 cancers. bioRxiv
9. Alexandrov, L.B. et al. (2015) Clock-like mutational processes in human somatic cells.
Nature
Genetics 47(12):1402-7.
10. Alexandrov, L.B. et al. (2016) Mutational signatures associated with tobacco smoking in human cancer.
Science 354(6312):618-622.
11. Ju, Y.S. et al. (2012) A transforming KIF5B and RET gene fusion in lung adenocarcinoma revealed from
whole-genome and transcriptome sequencing. Genome Research 22(3):436-45.
12. Zhang, C.Z. et al., (2015) Chromothripsis from DNA damage in micronuclei. Nature 522(7555):179-84.
첨부파일